PLS(部分的最小二乗法)の変数重要度をvip法を使って算出してみた
1. 背景
化学工学における物性値の予測やスペクトル解析においては、サンプル数に対して説明変数(特徴量)が非常に大きいということがある。そのような場合、一般的なOLS(最小二乗法)等の線形回帰モデルが使用できない。また、説明変数同士の相関が大きい(多重共線性、マルチコ)、適切な回帰モデルが構築できないという問題がある。このような問題に対して、PLS(部分的最小二乗法)が有効な手法である。PLSでは説明変数同士の相関をPCAにより排除し、マルチコを回避できる。また、PCAによりサンプル数が説明変数に比べ小さい場合でも回帰モデルが構築できる。また、目的変数との相関も考慮しながら、モデル が構築されるため、精度も期待できる。PLSはスモールデータ解析においてよくみられる手法の一つである。また、データ解析時には、説明変数の重要度が示せると、モデルの解釈ができたり、重要度の高い変数だけを残して、再度、モデルを学習させることで、汎化性能を高めることが可能である場合がある。PLSにはPLS専用の変数重要度算出方法(PLS-vip法)が提案されている。今回は、PLS-vip法を実装してみたいと思う。
2. PLS(部分的最小二乗法)
手法概要
PLSの導出、PLS-vip法の詳細な説明は他に譲ります。ここでは簡単な説明だけさせて頂きます。
- 部分最小二乗法 (Partial Least Squares, PLS) は、複数の互いに関連する説明変数 (predictor variables) がある場合に、それらの説明変数を少数の線形結合によって表現することで、目的変数 (response variable) の予測を行う回帰分析手法です。
- PLSは、主成分分析 (PCA) と回帰分析を組み合わせた手法であり、まず最初に説明変数の共分散行列を分解し、主成分を抽出します。次に、目的変数との共分散が大きい主成分を抽出し、それを基に部分的な回帰係数を求めます。この回帰係数を使って説明変数を線形結合することで、目的変数の予測を行います。
- PLSは、説明変数が多いデータセットや、説明変数同士の相関が高い場合に有用です。また、変数選択や特徴量抽出を行わずに、説明変数の情報を最大限に利用できるため、データ解析の精度を向上させることができます。
PLS-vip法(特徴量重要度)
- PLS-VIP (Variable Importance in Projection)法は、部分最小二乗法 (PLS)を用いた回帰分析において、各説明変数が目的変数に与える影響度を評価する方法の一つです。
- PLS-VIP法では、各説明変数の重要度を、それぞれの説明変数がPLSの射影に与える寄与度によって評価します。すなわち、PLSの射影において、各説明変数がどの程度目的変数との関連性があるかを測定し、その寄与度を基に、説明変数の重要度を算出します。重要度が高い説明変数ほど、目的変数に対する影響度が大きいと判断されます。
3. やりたいこと
こちらの水溶解度データセット(1290条件のSMILESデータとそれに対応する水溶解度logSを使用して、記述子から水溶解度logSを回帰するPLSモデルを構築し、PLS-vip法によって特徴量重要度を算出すること。
4. PLS-vip法により水溶解度予測モデルにおける各記述子の重要度を求めてみる。
4.1 データの準備
使用するデータを落としてきます。
#水溶解度データセット(SMILES)をスクレイピング
import urllib.request # URLで指定したファイルをダウンロードするライブラリ
url = 'http://modem.ucsd.edu/adme/data/databases/logS/data_set.dat'
urllib.request.urlretrieve(url, 'water_solubility.txt') データを整形します。
import pandas as pd
dataset = pd.read_csv('water_solubility.txt', sep='\t', header=None) # データの読み込み
dataset = dataset.drop([1],axis=1)
dataset.columns = ['SMILES', 'logS']
dataset.head()
rdkitを使用して、SMILESデータを記述子に変換します。
import pandas as pd
from rdkit import Chem
from rdkit.Chem import Descriptors
from rdkit.ML.Descriptors import MoleculeDescriptors
# 計算する記述子名の取得
descriptor_names = []
for descriptor_information in Descriptors.descList:
descriptor_names.append(descriptor_information[0])
print('計算する記述子の数 :', len(descriptor_names))
#記述子の計算
descriptor_calculator = MoleculeDescriptors.MolecularDescriptorCalculator(descriptor_names)
descriptors = [] # ここに計算された記述子の値を追加
for index, smiles_i in enumerate(smiles):
print(index + 1, '/', len(smiles))
molecule = Chem.MolFromSmiles(smiles_i)
descriptors.append(descriptor_calculator.CalcDescriptors(molecule))
descriptors = pd.DataFrame(descriptors, index=dataset.index, columns=descriptor_names)
if dataset.shape[1] > 1:
descriptors = pd.concat([y, descriptors], axis=1) # y と記述子を結合
#欠損値を含むデータがあるので削除
descriptors = descriptors.dropna(how='any')
学習データとバリデーションデータに分割します。
#説明変数と目的変数に分ける
X = descriptors.iloc[:,1:]
y = descriptors.iloc[:, 0]
#標準偏差が0の記述子を削除する
del_descriptors = X.columns[X.std() == 0]
X = X.drop(del_descriptors, axis=1)
#オートスケーリング
X_scaled = (X - X.mean()) / X.std()
y_scaled = (y - y.mean()) / y.std()
4.2 PLSモデルの構築
今回、PLSモデルのハイパーパラメーターである説明変数Xの潜在空間上でのサイズ(n_component)は54に設定した。
from sklearn.model_selection import train_test_split
from sklearn.cross_decomposition import PLSRegression
from sklearn.metrics import r2_score, mean_squared_error
import numpy as np
#学習データとテストデータに分割する
X_train, X_val, y_train, y_val = train_test_split(X_scaled, y_scaled, test_size=0.2, random_state=5, shuffle=True)
#plsの圧縮サイズを指定する
n_component = 54
pls = PLSRegression(n_components=n_component)
pls.fit(X_train, y_train.values.reshape(-1))
pred_train = pls.predict(X_train)
pred_val = pls.predict(X_val)
print('R2_score_train', r2_score(pred_train, y_train))
print('R2_score_val', r2_score(pred_val, y_val))
print('RMSE_train', np.sqrt(mean_squared_error(pred_train, y_train)))
print('RMSE_val', np.sqrt(mean_squared_error(pred_val, y_val)))4.3 特徴量重要度の可視化(PLS-vip)
続いて以下のスクリプトでpls-vip値を算出する関数を定義する。scikit-learnでplsモデルを学習させた後に得られる重み行列(x_weights_),目的変数yに対するローディング行列y_loadings_及び説明変数Xの潜在空間での値(x_scores_)を用いて算出できる。
#PLS-vop値を算出する関数
def pls_vip(pls_model):
W = pls_model.x_weights_
D = pls_model.y_loadings_
T = pls_model.x_scores_
M, R = W.shape
weight = np.zeros([R])
vips = np.zeros([M])
ssy = np.diag(T.T @ T @ D.T @ D)
total_ssy = np.sum(ssy)
for m in range(M):
for r in range(R):
weight[r] = np.array([(W[m, r]/ np.linalg.norm(W[:, r]))**2])
vips[m] = np.sqrt(M*(ssy @ weight) / total_ssy)
sel_var = np.arange(M)
#vip値が1よりも大きい特徴量名前を抽出する
sel_var = sel_var[vips>=1]
return sel_var, vips
上記の関数を使用してpls-vip値を算出して特徴量重要度を値が大きい順に並べて可視化します。
sel_var , vips = pls_vip(pls)
#データフレーム化
vips_df = pd.DataFrame(vips, index=X.columns, columns=['vips'])
vips_df = vips_df.sort_values('vips', ascending=True)
#可視化
import seaborn as sns
sns.set()
plt.figure(figsize=(20, 40))
plt.barh(vips_df.index, vips_df['vips'], align='center')
plt.yticks(fontsize=14)
plt.xticks(fontsize=14)
plt.title('Feature importance (PLS-vip)',fontsize=20)
plt.show()
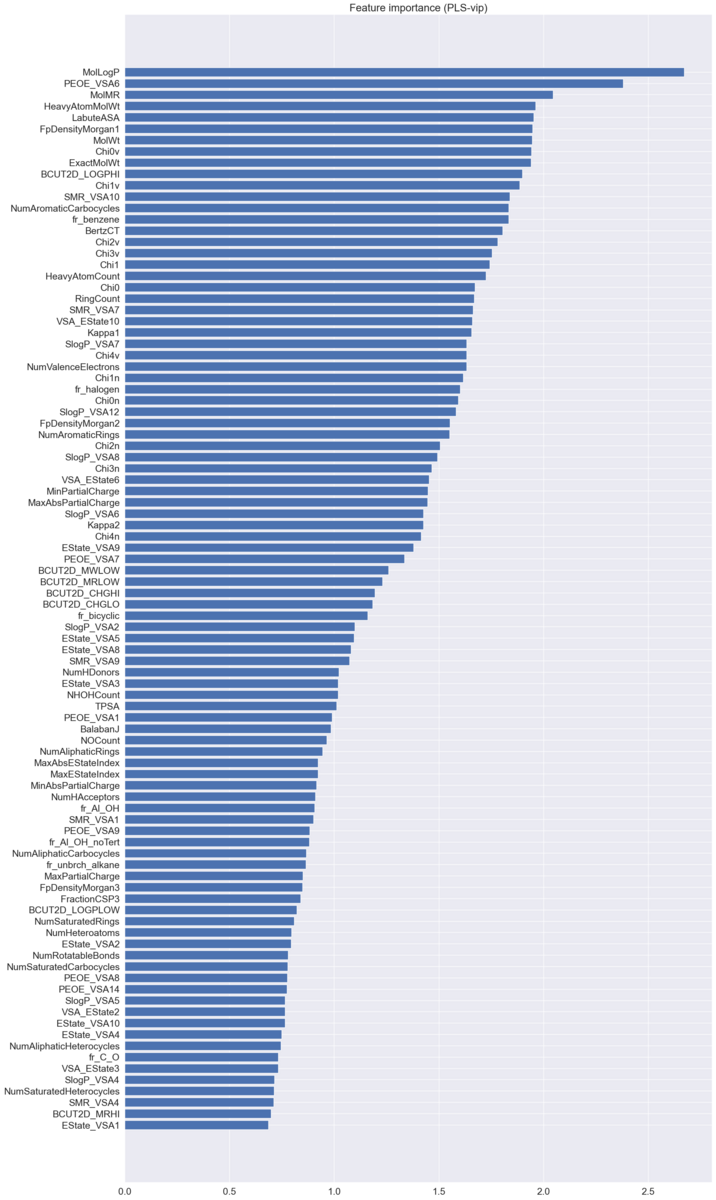
ちなみに、最も重要度の高いMolLogPは水/オクタノール分配係数(水溶性・脂溶性の指標)なので、水溶解度への寄与が大きいことは妥当な結果であると考えられます。
参考
こちらの本でPLSの詳細な導出やpls-vip法の説明が書かれています。参考にしてみてください。
