PytorchでPINNs(Physics Informed Neural Network)を実装してみる

0.目次
1.背景
業務でPINNs(Physics Informed Neural Network)を使用したいと思い、自身で実装する方法をまとめましたので備忘録がてらまとめてみます。
2.やりたいこと
1次元の自由振動系に対して振動するマスの変位を予測するPINNsを実装し、微分方程式を解くことにより得られる解析解及びデータドリブンに学習させたNNモデルと結果を比較してみます。
3.PINNs(Physics Informed Neural Network)
3.1.PINNsとは
PINNsはPhysics Informed Neural Networkの略称で物理法則を考慮したニューラルネットワークのことです。通常のニューラルネットワークではデータドリブンで学習を行うため、学習サイクルの中で学習データをもとに予測に重要な特徴を見つけたり、特徴量を生成することが行われます。通常のニューラルネットワークでは、学習データのみを頼りにするので、学習データ数が乏しい場合、思ったような予測精度が出なかったり、過学習をしてしまったりと課題があります。学習データ以外にそのタスクに関わるドメイン知識をニューラルネットワークに与えることはできないでしょうか?
PINNsではニューラルネットワークにドメイン知識を与えることが可能です。PINNsでは予めわかっているドメイン知識(物理式、支配方程式)を学習の中に入れ込むことができます。一般的なニューラルネットワークの学習時には逐次の予測値と教師データの差分を損失として与え、この損失を誤差逆伝播法により損失に対する各重みの勾配情報を上流のネットワークに伝え、勾配に基づいて各重みを更新することで学習を行います。PINNsでは予測値と教師データの差分に加えて、物理式、支配方程式に対する損失も入れ込んで学習を行います。予め分かっている物理式を満足しているかどうかに対する正則を入れ込むことでデータが少ない場合でもデータが全くない領域に対するモデルの精度も担保できます。学習データを再現するニューラルネットワークの重みの組み合わせは複数あるが、損失が予測値と教師データの差分のみであると手元にあるデータのみに適合すればよく、学習データ領域外のデータには関心がありません。PINNsにより支配方程式を損失に入れ込むことで学習データを再現する重みの組み合わせのうちの中から学習データにはない入力条件に対する現状のニューラルネットワークの重みでの支配方程式への当てはまりを考慮しながら学習(各重みを更新)するため、学習データ領域外の条件に対しても精度が担保することができます。
PINNsの工学分野での適用シーンを考えてみます。振動工学の分野においては、振動系に対して構造物(マス)の変位を予測したり、振動の減衰の様子を把握したい場面があるかと思います。例えば、1次のマス-バネ-ダンパーの自由振動系は以下の式で表されます。
ここで
は時間、
は変位、
はバネ定数、
はダンパー係数です。
1次の振動系であれば、微分方程式を解くことで簡単に解を求めることができるが、複雑な系であれば解を求めることは難しい。PINNsを使えば、初期の計測データを用いて学習を行うことで解を容易に予測することが可能である。
流体のシミュレーションにおいては、任意の時刻の流れ場を計算(予測)する際に、計算の縮退化ができる。流体の計算には一般的に大規模な計算格子を使用し、各格子点の物理量(速度、圧力)の時間発展を繰り返し計算により求めるため計算コストが高い。PINNsを学習させることにより時刻と格子座標を入力するとその時刻の物理量を瞬時に予測することができ、計算の高速化ができる。非圧縮の流体問題では、以下のような連続の式とNavier-Stokes式をNNの損失に組み込み、初期の各格子における物理量と境界条件を与えて学習を行うことで任意の時刻の物理量(速度、圧力)を予測できるモデルが作成できる。
ここで、
は密度、
は速度ベクトル、
は時間、
は勾配(空間微分演算子)、
は圧力、
は動粘性係数、
は重力ベクトルです。
下記はPINNsの火付け役になった論文です。こちらの論文でも流体の支配方程式をPINNsを用いて解法しています。
[https://arxiv.org/pdf/1711.10561.pdf:image=https://arxiv.org/pdf/1711.10561.pdf]
3.2.自動微分
上記の具体例で見てきたように物理式、支配方程式は時空間的な偏微分項を含む偏微分方程式です。PINNsでこれらの支配方程式を学習サイクルの中で満足しているかどうかをどのように計算/評価するのでしょうか?
ニューラルネットワークの学習では損失(教師の物理量と物理量の予測値)に対する各重みの勾配情報を微分の連鎖律を使って出力側から順次計算できる仕組みを使用しています。この微分の連鎖律(自動微分という)を使用して偏微分項の計算を高速に計算できます。(ただ、ネットワークを定義した段階ではネットワーク内の重みの勾配を求めることはできますが、入力変数に対する勾配(微分項)は計算できないので、入力変数もネットワークのグラフに組み込み、微分項を計算できるようにする設定が必要になります。)Pytorchなどの深層学習用のフレームワークではDefine by Runの仕組みが実装されており、順伝播の計算をしながらネットワークをグラフとして定義し、計算過程や各ノードの出力値、勾配情報を保存しています。これにより微分の連鎖律を使用したバックワードの高速な勾配計算(自動微分)を実現しています。順伝播型の勾配計算(数値微分)では何度も同じ計算が発生して計算効率が悪いですが、自動微分では計算グラフを構築しているため、一度の順方向計算と逆方向計算で勾配を効率的に求めることができます。
PytorchでPINNsを学習させる時に支配方程式(偏微分方程式)の微分項(勾配)を求める際はpytorchのautograd.gradを使用します。autograd.gradによって算出した微分項を用いて支配方程式を組み立てます。
import torch # 変数の定義 #勾配を計算するためにrequires_grad=Trueとする。 #PINNsの入力(1次の振動系であれば時間、流体問題であれば時間と格子座標) x = torch.tensor(2.0, requires_grad=True) # 関数の定義 #PINNsの出力 #実際は関数はNNの学習の中で決まっていく(NNの出力値、予測したい物理量) def function(x): return 3*x**2 + 2*x**3 # 変数に関する勾配の計算 #torch.autograd.gradを呼び出すことでグラフが作成され、勾配を計算できる。 #予測したい物理量に対する入力の勾配値(1次の振動系であれば、マスの変位に対する時間一階微分、流体であれば流速や圧力に対する時間あるいは空間一階微分) grad_x = torch.autograd.grad(function(x), x)[0] #支配方程式 #現時点の学習段階でのNNでの支配方程式への当てはまり度 loss = grad_x + c*x + ・・・
自動微分についてはこちらの記事を参考にしてください。 pystyle.info
4.マス-バネ-ダンパー系(1次の自由振動系)
以下の1次のマス-バネ-ダンパー系に対して解析結果とNNでデータのみを学習させた場合、PINNsで学習させた場合の結果を比較する。また、本記事ではマスの質量(=m)は1とする。
初期条件:
解析解:
ここで、 は振幅、
は初期位相です。
本記事の実装は下記の記事を参考にさせていただきました。
4.1.解析解
解析解を計算します。
def analytical_solution(g, w0, t): ''' g :ダンパーの減衰係数 / (2.0 * マスの質量) -> マス質量は1.0と仮定 w0 :周波数 t :np.linespace ''' assert g <= w0 w = np.sqrt(w0**2-g**2) phi = np.arctan(-g/w) A = 1/(2*np.cos(phi)) cos = np.cos(phi+w*t) sin = np.sin(phi+w*t) exp = np.exp(-g*t) x = exp*2*A*cos return x #パラメータ g, w0 = 2, 20 c, k = 2*g, w0**2 #time t = np.linspace(0,1,100) #x_analytical x_analytical = analytical_solution(g, w0, t) x_analytical = x_analytical.reshape(-1, 1)

4.2.NNによる解法
初期の計測データのみ(5点)を用いて、以降のマスの変位を再現できるか確認してみます。
まず時間を入力してその時刻に対応するマスの変位を予測するニューラルネットワーククラス(NNクラス)を定義します。
class NN(nn.Module): def __init__(self, n_input, n_output): super(NN, self).__init__() #set activation function self.activation = nn.Tanh() self.n_input = n_input self.n_output = n_output self.affine1 = nn.Linear(self.n_input, 50) self.affine2 = nn.Linear(50, 50) self.affine3 = nn.Linear(50, 50) self.affine4 = nn.Linear(50, self.n_output) def forward(self, t): t = self.activation(self.affine1(t)) t = self.activation(self.affine2(t)) t = self.activation(self.affine3(t)) t = self.affine4(t) return t
続いて、定義したNNクラスを使用してNNの学習/予測を行うクラスを定義します。
class NN_DataDriven(): def __init__(self, t, t_region, x): self.t = torch.tensor(t, requires_grad=True).float() self.t_region = torch.tensor(t_region, requires_grad=True).float() self.x = torch.tensor(x, requires_grad=True).float() #DEfine NN self.dnn = NN(1, 1) #optimizers self.optimizer = torch.optim.Adam(self.dnn.parameters(), lr=1e-3) def loss_func(self): x_pred = self.dnn(self.t) loss = torch.mean((self.x - x_pred)**2) return loss def train(self): epochs = 10000 for epoch in range(epochs): self.dnn.train() self.loss = self.loss_func() self.loss.backward() self.optimizer.step() self.optimizer.zero_grad() if epoch % 100 ==0: print( 'Iter %d, Loss: %.5e' % (epoch, self.loss.item())) def predict(self, t): t = torch.tensor(t, requires_grad=True).float() self.dnn.eval() x = self.dnn(t) return x
作成したNN_DataDrivenクラスについて説明します。クラスインスタント時に学習に使用する教師データのペアがある入力変数tと支配方程式への適合度を算出する際に使用する学習データでない入力変数t_regionと各時刻に対する変位の教師データxをTensor型で定義する。
loss_funcメソッドでは予測値と教師データ差を損失として定義している。
trainメソッドで、学習を行い、predictメソッドで入力時刻に対する変位を予測します。
NN_DataDrivenクラスをインスタンス化して、学習を行います。
t_pinn = np.linspace(0, 1, 100) t_pinn = t_pinn.reshape(-1, 1) # Pick up random data points random_list = [0, 5, 10, 20, 30] t_data = t_pinn[random_list] x_data = x_analytical[random_list] nn_datadriven = NN_DataDriven(t_data, t_pinn, x_data) nn_datadriven.train()
学習が完了したら、predictメソッドを使用して定義域全域で変位を予測し、解析解との比較を行なってみます。
#予測 x_pred = nn_datadriven.predict(t_pinn) x_pred = x_pred.detach().numpy() #プロット #予測値 plt.plot(t_pinn, x_pred,color='orange', lw=5,label='Predict') #解析解 plt.plot(t, x_analytical, label='Analytical') #学習データ plt.scatter(t_data, x_data,s=100,label='Train') plt.xlabel('Time') plt.ylabel('Displacement') plt.legend() plt.show()
 計測データのみの学習では、初期の計測データが存在する領域では精度良く予測できていますが、学習データ以降の時間域に対しては全く予測ができておらず、過学習に陥っていることが分かります。
計測データのみの学習では、初期の計測データが存在する領域では精度良く予測できていますが、学習データ以降の時間域に対しては全く予測ができておらず、過学習に陥っていることが分かります。
4.3.PINNsによる解法
PINNsによって初期の計測データからマスの変位の時間履歴(振動の様子)を予測するモデルを作成します。
学習に使用するデータは4.2.NNによる解法の場合と同じに設定します。
まず時間を入力してその時刻に対応するマスの変位を予測するニューラルネットワーククラス(NNクラス)を定義します。
class NN(nn.Module): def __init__(self, n_input, n_output): super(NN, self).__init__() #set activation function self.activation = nn.Tanh() self.n_input = n_input self.n_output = n_output self.affine1 = nn.Linear(self.n_input, 50) self.affine2 = nn.Linear(50, 50) self.affine3 = nn.Linear(50, 50) self.affine4 = nn.Linear(50, self.n_output) def forward(self, t): t = self.activation(self.affine1(t)) t = self.activation(self.affine2(t)) t = self.activation(self.affine3(t)) t = self.affine4(t) return t
続いて、定義したNNクラスを使用してPINNsのクラスを定義します。
class PINNs(): def __init__(self, t, t_region, x, c, k): self.c = c self.k = k self.t = torch.tensor(t, requires_grad=True).float() self.t_region = torch.tensor(t_region, requires_grad=True).float() self.x = torch.tensor(x, requires_grad=True).float() #DEfine NN self.dnn = NN(1, 1) #optimizers self.optimizer = torch.optim.Adam(self.dnn.parameters(), lr=1e-3) def net_f(self, t): x = self.dnn(t) x_t = torch.autograd.grad(x, t, grad_outputs=torch.ones_like(x),retain_graph=True, create_graph=True)[0] x_tt = torch.autograd.grad(x_t, t, grad_outputs=torch.ones_like(x_t),retain_graph=True, create_graph=True)[0] f = x_tt + self.c * x_t + self.k * x return f def loss_func(self): x_pred = self.dnn(self.t) f_pred = self.net_f(self.t_region) loss_x = torch.mean((self.x - x_pred)**2) loss_f = torch.mean(f_pred**2) loss = loss_x + loss_f*5e-4 return loss, loss_x, loss_f def train(self): epochs = 50000 for epoch in range(epochs): self.dnn.train() self.loss, self.loss_x, self.loss_f = self.loss_func() self.loss.backward() self.optimizer.step() self.optimizer.zero_grad() if epoch % 100 ==0: print( 'Iter %d, Loss: %.5e, Loss_x: %.5e, Loss_f: %.5e' % (epoch, self.loss.item(), self.loss_x.item(), self.loss_f.item())) def predict(self, t): t = torch.tensor(t, requires_grad=True).float() self.dnn.eval() x = self.dnn(t) f = self.net_f(t) return x, f
作成したPINNsクラスについて説明します。クラスインスタント時に学習に使用する教師データのペアがある入力変数tと支配方程式への適合度を算出する際に使用する学習データでない入力変数t_regionと各時刻に対する変位の教師データxをTensor型で定義する。この際、勾配を計算できるようにrequires_grad=Trueとしておく。
net_fメソッドでは、touch.autograd.gradを使用して、出力変数xに対する入力変数tの時間1階及び2階微分項を算出し、支配方程式の当てはまりを評価する。
loss_funcメソッドでは、出力値と教師データの差分にnet_fメソッドで算出した支配方程式への当てはまりを加えて、ネットワーク全体の損失を定義している。出力値と教師データの差分には入力と教師のペアがあるデータセットで評価し、支配方程式への当てはまりについてはモデル定義域を分割して得られた教師データがない入力条件を与えて評価しているがポイントである。これにより、学習データに存在していない
入力条件域に対しても支配方程式の当てはまりを考慮でき、定義域全域で精度が担保できる。
trainメソッドで、学習を行い、predictメソッドで入力時刻に対する変位を予測します。
PINNsクラスをインスタンス化して、学習を行います。
#モデルの定義域を分割(学習最中のモデルの支配方程式当てはまり度を算出する際に使用する) t_pinn = np.linspace(0, 1, 100) #NNモデルに入力できる形に変換 t_pinn = t_pinn.reshape(-1, 1) #t_pinnのうちが学習に使用するデータを5点抽出 random_list = [0, 5, 10, 20, 30] t_data = t_pinn[random_list] x_data = x_analytical[random_list] #インスタンス化 pinns = PINNs(t_data, t_pinn, x_data, c, k) #学習 pinns.train()
学習イタレーションを重ねるごとに損失全体、予測値と教師データの差分が小さく、支配方程式への適合度が改善していることが確認できます。
Iter 0, Loss: 4.13146e-01, Loss_x: 3.60471e-01, Loss_f: 1.05349e+02 Iter 100, Loss: 3.71868e-01, Loss_x: 3.68585e-01, Loss_f: 6.56660e+00 Iter 200, Loss: 3.70835e-01, Loss_x: 3.66550e-01, Loss_f: 8.56958e+00 Iter 300, Loss: 3.70310e-01, Loss_x: 3.65092e-01, Loss_f: 1.04367e+01 Iter 400, Loss: 3.69086e-01, Loss_x: 3.63777e-01, Loss_f: 1.06181e+01 Iter 500, Loss: 3.65856e-01, Loss_x: 3.58496e-01, Loss_f: 1.47206e+01 Iter 600, Loss: 3.62231e-01, Loss_x: 3.53761e-01, Loss_f: 1.69404e+01 Iter 700, Loss: 3.55470e-01, Loss_x: 3.42284e-01, Loss_f: 2.63724e+01 Iter 800, Loss: 3.44797e-01, Loss_x: 3.23800e-01, Loss_f: 4.19942e+01 Iter 900, Loss: 3.30014e-01, Loss_x: 3.00918e-01, Loss_f: 5.81928e+01 Iter 1000, Loss: 3.19377e-01, Loss_x: 2.80325e-01, Loss_f: 7.81050e+01 Iter 1100, Loss: 3.06024e-01, Loss_x: 2.59271e-01, Loss_f: 9.35068e+01 Iter 1200, Loss: 2.70418e-01, Loss_x: 2.19652e-01, Loss_f: 1.01531e+02 Iter 1300, Loss: 2.46888e-01, Loss_x: 1.84986e-01, Loss_f: 1.23804e+02 Iter 1400, Loss: 2.32732e-01, Loss_x: 1.67169e-01, Loss_f: 1.31126e+02 Iter 1500, Loss: 2.21095e-01, Loss_x: 1.50208e-01, Loss_f: 1.41774e+02 Iter 1600, Loss: 2.43834e-01, Loss_x: 1.38377e-01, Loss_f: 2.10915e+02 Iter 1700, Loss: 1.99829e-01, Loss_x: 1.25502e-01, Loss_f: 1.48655e+02 Iter 1800, Loss: 1.83171e-01, Loss_x: 1.09754e-01, Loss_f: 1.46834e+02 Iter 1900, Loss: 1.68960e-01, Loss_x: 9.84287e-02, Loss_f: 1.41062e+02 Iter 2000, Loss: 1.53515e-01, Loss_x: 8.57715e-02, Loss_f: 1.35486e+02 Iter 2100, Loss: 1.39630e-01, Loss_x: 7.56336e-02, Loss_f: 1.27992e+02 Iter 2200, Loss: 1.21700e-01, Loss_x: 6.35049e-02, Loss_f: 1.16391e+02 Iter 2300, Loss: 1.04604e-01, Loss_x: 5.25336e-02, Loss_f: 1.04141e+02 Iter 2400, Loss: 8.70835e-02, Loss_x: 3.99109e-02, Loss_f: 9.43451e+01 ... Iter 23000, Loss: 9.58961e-04, Loss_x: 1.96658e-05, Loss_f: 1.87859e+00 Iter 23100, Loss: 7.20585e-04, Loss_x: 1.42954e-05, Loss_f: 1.41258e+00 Iter 23200, Loss: 7.42390e-04, Loss_x: 1.98486e-05, Loss_f: 1.44508e+00 Iter 23300, Loss: 2.59901e-03, Loss_x: 6.43220e-05, Loss_f: 5.06938e+00
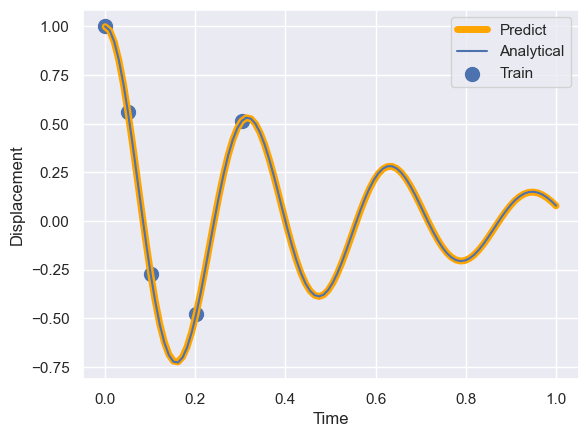
学習が完了したので、predictメソッドを使用して定義域全域で変位を予測し、解析解との比較を行なってみます。
#予測 x_pred, f_pred = pinns.predict(t_pinn) x_pred = x_pred.detach().numpy() #プロット #予測値 plt.plot(t_pinn, x_pred, lw=5,label='Predict') #解析解 plt.plot(t, x_analytical, label='Analytical') #学習データ plt.scatter(t_data, x_data,s=100,label='Train') plt.xlabel('Time') plt.ylabel('Displacement') plt.legend() plt.show()

学習データがない領域に対しても高精度に予測ができています。
5.まとめ
今回、マス-バネ-ダンパーの一次の振動系に着目してpytorchを使用してPINNsを実装してみました。予め支配方程式がわかっていれば、初期の少数のサンプルで学習データ条件外の条件に対しても高精度に予測できるPINNsの強力さを体感できました。実務でもこの実装経験をいきたいと思います。
AttentionモデルとCNNモデルの判断根拠を比較してみる

0.目次
1.やりたいこと
・sin波とcos波の分類を行う機械学習モデルを作成し、その予測の判断根拠を可視化する。
・分類を行う機械学習モデルは、Self-AttentionモデルとCNNモデルを作成する。Self-Attentionモデルではattention-weightを可視化することで、CNNモデルではGradCAMを使用することで可視化し、モデル毎の判断根拠を比較する。
2.使用するライブラリ
import numpy as np import matplotlib.pyplot as plt from torch.utils.data import DataLoader import torch import torch.optim as optim import torch.nn as nn import torch.nn.functional as F from torch.autograd import Function import seaborn as sns sns.set()
3.使用するデータ
sin波とcos波にノイズを加えたデータをそれぞれ100個作成し、sin波であれば[1, 0]のラベルをcon波であれば[0, 1]のラベルを付加します。
class Dataset: def __init__(self, sequential_length=100, num_point=100): # ノイズを加えたsin波とcos波のデータ生成 np.random.seed(42) # 乱数シードの設定 self.sequential_length=sequential_length self.num_point = num_point # sin波にノイズを加えたデータの生成 sin_data = np.sin(np.linspace(0, 2 * np.pi, self.num_point)) # sin波の生成 sin_noisy_data = [] for _ in range(self.sequential_length): noisy_data = sin_data + np.random.normal(0, 0.1, size=sin_data.shape) # ノイズの追加 sin_noisy_data.append(noisy_data) sin_noisy_data = np.concatenate(sin_noisy_data) sin_noisy_data = sin_noisy_data.reshape(self.sequential_length, -1) # cos波にノイズを加えたデータの生成 cos_data = np.cos(np.linspace(0, 2 * np.pi, self.num_point)) # cos波の生成 cos_noisy_data = [] for _ in range(self.sequential_length): noisy_data = cos_data + np.random.normal(0, 0.1, size=cos_data.shape) # ノイズの追加 cos_noisy_data.append(noisy_data) cos_noisy_data = np.concatenate(cos_noisy_data) cos_noisy_data = cos_noisy_data.reshape(self.sequential_length, -1) # データの結合とシャッフル data = np.concatenate((sin_noisy_data, cos_noisy_data)) data = data.reshape(2 * self.sequential_length, -1, 1) sin_label1 = np.ones((self.sequential_length, 1)) sin_label2 = np.zeros((self.sequential_length, 1)) sin_label = np.concatenate((sin_label1, sin_label2), axis=1) cos_label1 = np.zeros((self.sequential_length, 1)) cos_label2 = np.ones((self.sequential_length, 1)) cos_label = np.concatenate((cos_label1, cos_label2), axis=1) labels = np.concatenate((sin_label, cos_label)) #torchに変換する #[batch_size, 1, sequential_length] self.X = torch.tensor(data, dtype=torch.float32).transpose(1,2) self.t = torch.tensor(labels, dtype=torch.float32) def __len__(self): return len(self.X) def __getitem__(self, index): return self.X[index], self.t[index]
以下のようなデータが生成されます。

4.Self-Attentionモデルの学習/判断根拠の可視化
4.1.Self-Attention
まず、Attention(注意機構)について簡単に解説します。(※詳細な解説は他の記事に譲ります。)Attentionは自然言語処理の分類や翻訳のタスクにおいて、単語間の関連付け、翻訳元と翻訳先の単語との関連付けを動的に行うことで入力情報を全体を見渡して重要な情報を抽出することが可能で自然言語処理や画像分類のタスクに変革をもたらしました。自然言語処理のタスクにおいては、それまでのLSTM等のRNNの欠点である並列計算ができない課題を克服しました。画像分類のタスクでは、CNNの欠点である離れた情報の関連付けを行うことが可能になり、画像分類の予測精度にも寄与しました。例えば、以下の画像のように位置的に離れた情報を関連付けることが出来ます。

Transfomerの中では、Self-Attention機構とSorceTarget-Attentionが実装され、現在注目されているChatGPTにも組み込まれてます。
以下にSelf-Attentionの仕組み/計算方法を簡単に示します。

系列データABC(何らかの文章と思ったら良い)をSelf-Attentionにより単語間の関連付けを行うことを考えます。次のステップで計算を行います。
1. 各単語を数値に変換するEmbeddingを行います。
2.Embeddingした各単語に対してさらにEmbeddingを行い、Query、Key、Valueを作成します。
3.QueryとKeyの内積を計算し、Queryに設定した単語(注目する単語、他の単語と関連付けした量を算出したい単語)とKeyに設定した単語の類似度を計算する。
4.類似度でValueを重みづけを行う。
5.重みづけしたValue'(Weighted-Value)をすべての単語に対して計算したの後に総和をとる。
以上のステップにより他の単語との関連付けを動的に行う。学習の中でEmbeddingレイヤーの重みが最適化され、分類精度を上げるのにどこに注目すればよいか(attentionすればよいか)を学習する。学習後に得られる各単語間の類似はAttentionWeightと呼ばれ、機械学習モデルの予測の判断根拠を可視化する際に有用である。AttentionはXAIの一面を持つことで説明性を付加してくれる。今回はこのAttentionWeightを用いて、判断根拠を可視化する。
こちらの記事を見ると、Attentionの仕組み、計算方法が良く分かると思います。 jalammar.github.io
4.2.学習モデルの定義
それでは、Sel-Attentionモデルを構築、学習を行い、Attention-weigthを可視化してみます。
まず、Self-Attentionモデルのアーキテクチャーを下記のように定義します。 入力データ(sin波、cos波)をEmbbedingします。その後、Self-Attentionに使用するため、Query, Key, ValueをEmbeddingを行い作成します。作成したQuery, Key, Valueを使用してSelf-Attention計算を行い、最後にFlatten処理を行い、Affineレイヤーを結合し、出力層につながる構造になっています。
class AttentionClasifier(nn.Module): def __init__(self, dim_emb:int=10, num_heads:int=1, sequential_length=100): super(Clasifier, self).__init__() #埋め込み次元 self.dim_emb = dim_emb #アテンションヘッドの数 self.num_heads = num_heads #各ヘッドの次元数 self.head_dim = dim_emb // num_heads # self.sqrt_dh = self.head_dim**0.5 #埋め込み self.emb = nn.Linear(1, dim_emb) #query埋め込み self.query = nn.Linear(dim_emb, dim_emb, bias=False) #key埋め込み self.key = nn.Linear(dim_emb, dim_emb, bias=False) #value埋め込み self.value = nn.Linear(dim_emb, dim_emb, bias=False) # self.w_o = nn.Linear(dim_emb, dim_emb) #平坦化 self.flatten = nn.Flatten() # self.affine = nn.Linear(sequential_length * dim_emb, 2) def forward(self, x): batch_size, _ , sequence_length = x.size() #形状変換 #(batch_size, 1, sequence_length) -> (batch_size, sequence_length, 1) x = x.transpose(1,2) #埋め込み ## (batch_size, sequence_length, 1) -> (batch_size, sequence_length, dim_emb) x = self.emb(x) #query, key, value ## (batch_size, sequence_length, dim_emb) -> (batch_size, sequence_length, dim_emb) q = self.query(x) k = self.key(x) v = self.value(x) #q, k, vをヘッドに分ける ## (batch_size, sequence_length, dim_emb) -> (batch_size, sequence_length, num_heads, dim_emb//num_heads) q = q.view(batch_size, sequence_length, self.num_heads, self.head_dim) k = k.view(batch_size, sequence_length, self.num_heads, self.head_dim) v = v.view(batch_size, sequence_length, self.num_heads, self.head_dim) #セルフアテンションができるように変換 ## (batch_size, sequence_length, num_heads, dim_emb//num_heads) -> (batch_size, num_heads, sequential_length, dim_emb//num_heads) q = q.transpose(1,2) k = k.transpose(1,2) v = v.transpose(1,2) #内積 ## (batch_size, num_heads, sequential_length, dim_emb//num_heads) -> (batch_size, num_heads, dim_emb//num_heads, sequential_length) k_T = k.transpose(2,3) ## (batch_size, num_heads, sequential_length, dim_emb//num_heads) * (batch_size, num_heads, dim_emb//num_heads, sequential_length) -> (batch_size, num_heads, sequential_length, seqential_length) dots = (q @k_T) / self.sqrt_dh #列方向にソフトマックス attn = F.softmax(dots, dim=-1) #加重和 ## (batch_size, num_heads, sequential_lenght, sequential_length) * (batch_size, num_heads, sequential_length, dim_emb//num_heads) -> (batch_size, num_heads, sequential_length, dim_emb//num_heads) out = attn @ v ## (batch_size, num_heads, sequential_length, dim_emb//num_heads) -> (batch_size, sequential_lengh, num_heads, dim_emb//num_heads) out = out.transpose(1,2) ## (batch_size, sequential_lengh, num_heads, dim_emb//num_heads) -> (batch_size, sequential_lengh, dim_emb) out = out.reshape(batch_size, sequence_length, self.dim_emb) out = self.w_o(out) out = self.flatten(out) out = self.affine(out) return out, attn
4.3.Dataloaderの作成/学習
続いて、PytorchのDataloaderを使用してバッチ学習用にデータを作成し、学習モデルアーキテクチャーをインスタンス化します。その後、学習を行います。
#データセットの作成 dataset = Dataset() #バッチサイズ batch_size = 8 #バッチ学習のためにDataloaderに変換する loader = DataLoader(dataset=dataset, batch_size=batch_size, shuffle=True) #学習モデルをインスタンス化 cls_attr= AttentionClasifier() # 損失関数と最適化手法の定義 learning_rate = 1e-3 num_epochs = 1000 criterion = nn.CrossEntropyLoss() optimizer = optim.Adam(cls_attr.parameters(), lr=learning_rate) # 学習ループ total_step = len(loader) for epoch in range(num_epochs): for i, (data, labels) in enumerate(loader): # フォワードパス outputs, attn = cls_attr(data) loss = criterion(outputs, labels) # バックワードパスと最適化 optimizer.zero_grad() loss.backward() optimizer.step() if (i + 1) % 10 == 0: print(f"Epoch [{epoch + 1}/{num_epochs}], Step [{i + 1}/{total_step}], Loss: {loss.item():.4f}")
学習を行うと、lossが更新されます。lossが最終的に0になり、完全に分類されます。
Epoch [1/1000], Step [10/25], Loss: 0.7461 Epoch [1/1000], Step [20/25], Loss: 0.6778 Epoch [2/1000], Step [10/25], Loss: 0.5947 Epoch [2/1000], Step [20/25], Loss: 0.4390 Epoch [3/1000], Step [10/25], Loss: 0.0641 Epoch [3/1000], Step [20/25], Loss: 0.0084 Epoch [4/1000], Step [10/25], Loss: 0.0014 Epoch [4/1000], Step [20/25], Loss: 0.0008 Epoch [5/1000], Step [10/25], Loss: 0.0005 Epoch [5/1000], Step [20/25], Loss: 0.0005 Epoch [6/1000], Step [10/25], Loss: 0.0004 Epoch [6/1000], Step [20/25], Loss: 0.0004 Epoch [7/1000], Step [10/25], Loss: 0.0003 Epoch [7/1000], Step [20/25], Loss: 0.0003 Epoch [8/1000], Step [10/25], Loss: 0.0003 Epoch [8/1000], Step [20/25], Loss: 0.0002 Epoch [9/1000], Step [10/25], Loss: 0.0002 Epoch [9/1000], Step [20/25], Loss: 0.0002 Epoch [10/1000], Step [10/25], Loss: 0.0002 Epoch [10/1000], Step [20/25], Loss: 0.0002 Epoch [11/1000], Step [10/25], Loss: 0.0002 Epoch [11/1000], Step [20/25], Loss: 0.0001 Epoch [12/1000], Step [10/25], Loss: 0.0001 Epoch [12/1000], Step [20/25], Loss: 0.0002 Epoch [13/1000], Step [10/25], Loss: 0.0001 ... Epoch [999/1000], Step [10/25], Loss: -0.0000 Epoch [999/1000], Step [20/25], Loss: -0.0000 Epoch [1000/1000], Step [10/25], Loss: -0.0000 Epoch [1000/1000], Step [20/25], Loss: -0.0000
4.4.Self-Attentionによる判断根拠の可視化
AttentionWeightを可視化してみます。
#データを取得 data_ = dataset.X #AttentionWeightを取得 cls_attr.eval() _, attn = cls_attr(data_) data_ = data_.reshape(-1, 100) #データ番号 case = 101 #プロット attn_ = attn[case].reshape(100, 100) plt.imshow(attn_.detach().numpy()) plt.show()
以下にcos波とsin波のAttentionMapの比較を示します。
 学習の中で波の山の位置と谷の位置に関連があるという情報を獲得しています。面白いですね。Attentionmapと波形を重ねてみます。
学習の中で波の山の位置と谷の位置に関連があるという情報を獲得しています。面白いですね。Attentionmapと波形を重ねてみます。
#データ data_ = dataset.X #AttentionWeight cls_attr.eval() _, attn = cls_attr(data_) data_ = data_.reshape(-1, 100) #データ番号 case = 1 #プロット fig, ax = plt.subplots() # カラーマップを作成 cmap = plt.get_cmap('Blues') attn_ = attn[case].reshape(100, 100) attn_list = attn_.detach().numpy().sum(axis=0) attn_list = (attn_list - attn_list.min()) / (attn_list.max() - attn_list.min()) attn_list = attn_list.tolist() #AttentionWeightで色付け for i in range(100): ax.axvspan(xmin=i, xmax=i+1, color=cmap(attn_list[i]), alpha=0.25) plt.plot(np.linspace(0, 100, 100), data_[case]) plt.show()
 Self-Attentionモデルは山と谷の位置を見て予測をしていることが分かります。
Self-Attentionモデルは山と谷の位置を見て予測をしていることが分かります。
5.CNNモデルの学習/判断根拠の可視化
5.1.GradCAM
GradCAM(Gradient-weighted Class Activation Mapping)は、畳み込みニューラルネットワーク(CNN)を用いた画像認識タスクにおいて、ネットワークの予測結果を可視化するための手法です。GradCAMは、モデルが画像内のどの領域に注目して予測を行ったのかを視覚的に理解するために使用されます。GradCAMの処理手順は以下のようになります。
1.まず、対象となる画像をネットワークに入力し、予測結果を得ます。
2.ネットワークの最終畳み込み層の出力(特徴マップ)に対して、予測結果に対する勾配を求めます。勾配は、予測クラスに対する特徴マップの各ピクセルの重要度を表します。
3.勾配を特徴マップの各チャンネルに適用し、チャンネルごとに重要度を求めます。この重要度は、各チャンネルが予測にどれだけ寄与しているかを示します。
4.チャンネルごとの重要度を特徴マップに重み付けして足し合わせ、GradCAMマップを生成します。GradCAMマップは、予測クラスに対して重要な領域を強調したヒートマップとして表現されます。
5.GradCAMマップを元の入力画像のサイズにアップサンプリングします。
6.最後に、GradCAMマップを元の入力画像に重ね合わせることで、モデルが予測に使用した重要な領域を可視化します。
このようにして生成されたGradCAMマップは、ネットワークの予測の根拠を可視化するために利用され、画像認識モデルの解釈性や信頼性を向上させるのに役立ちます。
5.2.学習モデルの定義
一般的な1次元のCNNモデルを定義します。
class Conv1dClasifier(nn.Module): def __init__(self, num_filter, kernel_size): super(Conv1dClasifier, self).__init__() self.num_filter = num_filter self.kernel_size = kernel_size self.conv1 = nn.Conv1d(1, num_filter, kernel_size) self.pool1 = nn.MaxPool1d(2) self.conv2 = nn.Conv1d(num_filter, num_filter*2, kernel_size) self.pool2 = nn.MaxPool1d(2) self.flatten = nn.Flatten() self.affine1 = nn.Linear(368, 100) self.affine2 = nn.Linear(100, 10) self.affine3 = nn.Linear(10, 2) def forward(self, x): x = self.pool1(F.relu(self.conv1(x))) x = self.pool2(F.relu(self.conv2(x))) x = self.flatten(x) x = F.relu(self.affine1(x)) x = F.relu(self.affine2(x)) x = self.affine3(x) return x
5.3.Dataloaderの作成/学習
#学習及び評価 #データセットの作成 dataset = Dataset() #バッチサイズ batch_size = 8 #バッチ学習のためにDataloaderに変換する loader = DataLoader(dataset=dataset, batch_size=batch_size, shuffle=True) #学習モデルをインスタンス化 cls = Conv1dClasifier(num_filter=8, kernel_size=3) # 損失関数と最適化手法の定義 learning_rate = 1e-3 num_epochs = 1000 criterion = nn.CrossEntropyLoss() optimizer = optim.Adam(cls.parameters(), lr=learning_rate) # 学習ループ total_step = len(loader) for epoch in range(num_epochs): for i, (data, labels) in enumerate(loader): # フォワードパス outputs = cls(data) loss = criterion(outputs, labels) # バックワードパスと最適化 optimizer.zero_grad() loss.backward() optimizer.step() if (i + 1) % 10 == 0: print(f"Epoch [{epoch + 1}/{num_epochs}], Step [{i + 1}/{total_step}], Loss: {loss.item():.4f}")
学習を行うと、lossが更新されます。lossが最終的に0になり、完全に分類されます。
Epoch [1/1000], Step [10/25], Loss: 0.6089 Epoch [1/1000], Step [20/25], Loss: 0.4347 Epoch [2/1000], Step [10/25], Loss: 0.4945 Epoch [2/1000], Step [20/25], Loss: 0.1950 Epoch [3/1000], Step [10/25], Loss: 0.5618 Epoch [3/1000], Step [20/25], Loss: 0.1849 Epoch [4/1000], Step [10/25], Loss: 0.4518 Epoch [4/1000], Step [20/25], Loss: 0.2676 Epoch [5/1000], Step [10/25], Loss: 0.3489 Epoch [5/1000], Step [20/25], Loss: 0.2580 Epoch [6/1000], Step [10/25], Loss: 0.2527 Epoch [6/1000], Step [20/25], Loss: 0.1663 Epoch [7/1000], Step [10/25], Loss: 0.3255 Epoch [7/1000], Step [20/25], Loss: 0.2407 Epoch [8/1000], Step [10/25], Loss: 0.1572 Epoch [8/1000], Step [20/25], Loss: 0.3102 Epoch [9/1000], Step [10/25], Loss: 0.3041 Epoch [9/1000], Step [20/25], Loss: 0.3000 Epoch [10/1000], Step [10/25], Loss: 0.3676 Epoch [10/1000], Step [20/25], Loss: 0.2177 Epoch [11/1000], Step [10/25], Loss: 0.3556 Epoch [11/1000], Step [20/25], Loss: 0.2806 Epoch [12/1000], Step [10/25], Loss: 0.1376 Epoch [12/1000], Step [20/25], Loss: 0.3395 Epoch [13/1000], Step [10/25], Loss: 0.2662 ... Epoch [999/1000], Step [10/25], Loss: -0.0000 Epoch [999/1000], Step [20/25], Loss: -0.0000 Epoch [1000/1000], Step [10/25], Loss: -0.0000 Epoch [1000/1000], Step [20/25], Loss: -0.0000
5.4.GradCAMによる判断根拠の可視化
GradCAMを行うクラスを定義します。
class GradCAM: def __init__(self, model, feature_layer): self.model = model self.feature_layer = feature_layer self.model.eval() self.feature_grad = None self.feature_map = None self.hooks = [] # 最終層逆伝播時の勾配を記録する def save_feature_grad(module, in_grad, out_grad): self.feature_grad = out_grad[0] self.hooks.append(self.feature_layer.register_backward_hook(save_feature_grad)) # 最終層の出力 Feature Map を記録する def save_feature_map(module, inp, outp): self.feature_map = outp[0] self.hooks.append(self.feature_layer.register_forward_hook(save_feature_map)) def forward(self, x): return self.model(x) def backward_on_target(self, output, target): self.model.zero_grad() one_hot_output = torch.zeros([1, output.size()[-1]]) print(one_hot_output.shape) one_hot_output[0][target] = 1 output.backward(gradient=one_hot_output, retain_graph=True) def clear_hook(self): for hook in self.hooks: hook.remove()
上記で定義したGradCAMクラスをインスタンス化し、cam値を算出します。
gradcam = GradCAM(cls, cls.conv2) model_output = gradcam.forward(dataset.X[0].unsqueeze(0)) target = model_output.argmax(1).item() gradcam.backward_on_target(model_output, target) # Get feature gradient feature_grad = gradcam.feature_grad.data.numpy()[0] # Get weights from gradient weights = np.mean(feature_grad, axis=(1)) # Take averages for each gradient # Get features outputs feature_map = gradcam.feature_map.data.numpy() gradcam.clear_hook() # Get cam cam = np.sum((weights * feature_map.T), axis=1).T cam = np.maximum(cam, 0) # apply ReLU to cam #リサイズ cam = np.interp(np.linspace(0,1, 100), np.linspace(0,1,47), cam) #正規化 cam = (cam - cam.min()) / (cam.max() - cam.min()) cam
GradCAMの実装はこちらを参考にしました。 tech.jxpress.net
cam値が求まったので、波形とcam値を重ねてみます。
#データ data_ = dataset.X.reshape(-1, 100) #データ番号 case = 101 fig, ax = plt.subplots() # カラーマップを作成 cmap = plt.get_cmap('Blues') for i in range(100): ax.axvspan(xmin=i, xmax=i+1, color=cmap(cam[i]), alpha=0.25) plt.plot(np.linspace(0, 100, 100), data_[case]) plt.show()
 Self-attentionモデルとは異なり、sin波、cos波に依らず、sin波の山と谷の位置に注目して判断していることが伺えます。CNNモデルでは注目する位置は変わっていませんが、Self-Attentionモデルは波形によって注目する位置が変わっていて、面白いですね。
Self-attentionモデルとは異なり、sin波、cos波に依らず、sin波の山と谷の位置に注目して判断していることが伺えます。CNNモデルでは注目する位置は変わっていませんが、Self-Attentionモデルは波形によって注目する位置が変わっていて、面白いですね。
6.まとめ
今回、Self-AttentionモデルとCNNモデルを作成し、sin波とcos波の分類を行い、その判断根拠の可視化と両モデルの判断根拠を比較してみました。Self-Attentionモデルでは学習の中で山と谷の位置に関係があるという情報を自動で獲得していて非常に興味深かったです。
7.参考
SnapshotPODについてまとめてみる
 出所:https://www.nagare.or.jp/download/noauth.html?d=30-2rensai2.pdf&dir=54
出所:https://www.nagare.or.jp/download/noauth.html?d=30-2rensai2.pdf&dir=54
0.はじめに
機械学習において、主成分分析はデータの可視化や次元圧縮等に使用される。
この主成分分析を流体の数値計算に適用すること(SnapshotPOD)で流れの空間モードの分解や数値計算の縮退化(高速化)が行える。
今回、SnapshotPODについて調べたので簡単にまとめてみる。
1.主成分分析(Principal Component Analysis)
機械学習の分野では、多次元データに対して変数間の分散が大きくなる方向にデータを射影することで低次元データに次元削減する目的で主成分分析が行われる。
低次元のデータにできると、(例えば2次元、3次元に縮退できると)データの可視化が行えたり、各射影空間での軸が直行することが保証されるのでマルチコ(多重共線性)の回避ができる。
具体的には、下記のように1次元目に条件数を並べ、2次元目に説明変数を並べたデータ行列をX(N×M)とする。
 このデータ行列の分散共分散行列に関する固有値問題を解くことで固有値と固有ベクトル行列が求まる。得られた固有ベクトル行列(固有値が大きい順に並び替え)とデータ行列とで行列積をとることで主成分得点が得られる。第1主成分はデータを最も分散が大きくなる軸に射影したデータ、第2主成分はそれに直行し、次に分散が大きくなる軸に射影したデータとなります。
このデータ行列の分散共分散行列に関する固有値問題を解くことで固有値と固有ベクトル行列が求まる。得られた固有ベクトル行列(固有値が大きい順に並び替え)とデータ行列とで行列積をとることで主成分得点が得られる。第1主成分はデータを最も分散が大きくなる軸に射影したデータ、第2主成分はそれに直行し、次に分散が大きくなる軸に射影したデータとなります。
 pythonでは固有値問題は以下のように実装できる。この例では固有値問題を解き、第二主成分までを使用して次元削減を行っている。
pythonでは固有値問題は以下のように実装できる。この例では固有値問題を解き、第二主成分までを使用して次元削減を行っている。
import numpy as np import numpy.linalg as LA #X.T@Xの固有値、固有ベクトルを求める sigma, U = LA.eig(X.T@X) #固有値が大きいものから並べ直し indices = np.argsort(-sigma) sigma = sigma[indices] #Uもsigmaに合わせて並べ直す U = U[:, indices] X_low = X_low @ U #指定した次元まで縮退化する n_components = 2 X_new = X_low[:n_components, :]
上記の実装例では固有値問題を解いたが、特異値分解(SVD)によっても主成分分析ができる。例えば、scikit-learn.decompositionのPCAは特異値分解(SVD)が実装されており、下記のコードで主成文分析ができる。
from sklearn.decomposition import PCA pca = PCA(n_components=2) X_new = pca.fit_transform(X)
この縮退化(次元削減)技術を流体の流れ場のモード分解に適用したものがSnapshotPODである。
2.SnapshotPOD
流体の計算では多数の計算格子を用意して支配方程式の時間発展を解かなければならないため、計算コストが一般的に高い。
SnapshotPODにより、流体の流れ場の時間発展データを時間軸方向に次元削減し、低次の空間モードを抽出することで計算コストを大幅に削減することができる。(後述)
以下では、2次元の流体計算を例にSnapshotPODを考えてみる。
まず、x座標、y座標の格子点での物理量を1つの縦ベクトルが1タイムステップを表すように変換する。
 この変換により、1次元目各時刻の計算格子全点での物理量(速度)、2次元目が時間になる。
このデータ行列をXとして固有値問題を解くことで固有値、固有ベクトル行列が求まる。
この変換により、1次元目各時刻の計算格子全点での物理量(速度)、2次元目が時間になる。
このデータ行列をXとして固有値問題を解くことで固有値、固有ベクトル行列が求まる。
ここで得られた固有値、固有ベクトル及びデータ行列Xを使用して空間モード(POD基底という)は以下のように求まる。
※一般的にはデータ行列から時間平均流れ場を引き算したデータ行列の固有値問題を解く。
Pythonでは以下のように実装できる。
import numpy as np import numpy.linalg as LA #二次元解析データ(N1, N2, M)を(N1+N2, M)に変換 img_data = img_data.reshape(-1, N1*N2).T #snapshot POD sigma, U = LA.eig(img_data.T@img_data) #X.T@Xの固有値、固有ベクトルを求める sigma = np.sqrt(sigma2) sigma = np.nan_to_num(sigma) #Nanが発生していたので、0に置き換え indices = np.argsort(-sigma) #固有値が大きいものから並べ直し sigma = sigma[indices] U = U[:, indices] #Uもsigmaに合わせて並べ直す sigma_inv = np.diag(1/sigma) #∑-1を計算する V = img_data_mat_l @ U @ sigma_inv #V = X @ U @ ∑-1で計算できる #空間モードを抽出する mode = 1 V[mode, :]
 POD基底は固有値の大きいモードほど、空間的に大きな構造をもつ。
例えば、下の例では1次モード(上から順に固有値が大きい)が最も空間的に大きな渦構造をもつモードが抽出されてる。
固有値が小さくなるほど、小さな渦構造を示している。
POD基底は固有値の大きいモードほど、空間的に大きな構造をもつ。
例えば、下の例では1次モード(上から順に固有値が大きい)が最も空間的に大きな渦構造をもつモードが抽出されてる。
固有値が小さくなるほど、小さな渦構造を示している。
 このようにSnapshotPODにより、時間発展する流れ場に対して空間モードが抽出でき、流れを支配する空間構造を把握できる。
このようにSnapshotPODにより、時間発展する流れ場に対して空間モードが抽出でき、流れを支配する空間構造を把握できる。
さらに、SnapshotPODを使用することで流体解析の縮退化ができる。各空間モードに対して時間的に変化する係数をかけたものの足し合わせで流れ場を表現できると考えられる。(波の重ね合わせ、モードの重ね合わせ)
 これによりR(<<M)までのモードと時間に依存する係数の和で流れを再構築できる。CFDで計算する場合、空間に離散化した(N点)の時間発展を解かなければならないが、SnapshotPODを使用すると、R(<<N)次元の各モードの係数の時間発展を解けばよくなり、大幅に計算コストを削減できる。
これによりR(<<M)までのモードと時間に依存する係数の和で流れを再構築できる。CFDで計算する場合、空間に離散化した(N点)の時間発展を解かなければならないが、SnapshotPODを使用すると、R(<<N)次元の各モードの係数の時間発展を解けばよくなり、大幅に計算コストを削減できる。

GCN(Graph Convolutional Networks)を使用して化合物の物性値を予測してみた

0.はじめに
化合物の物性予測の際には、記述子を用いたり、フィンガープリントを用いたり等アプローチがいくつかあります。 今回、化合物の構造データ(グラフデータ)から畳み込み演算により特徴を抽出しながらモデルを構築するGCNN(Graph Convolutional Neural Network)を使用して化合物の水溶解度予測の実装をしてみました。 実装には、Pytorch Geometricを使用しました。インストール方法等に関しての説明は割愛させて頂きます。
masao-ds.hatenablog.com こちらの記事では記述子を使用して同様の化合物データの水溶解度予測をしています。
1.やりたいこと
化合物の構造データ(グラフデータ)を入力してその化合物の水溶解度を予測するモデルを構築します。

2.使用するデータセット及び形式
こちら の水溶解度データを使用します。ダウンロードしたデータを下記のようなcsv形式に変換します。
| No | SMILES | logS |
|---|---|---|
| sample_1 | CC(N)=O | 1.58 |
| sample_2 | CNN | 1.34 |
| sample_3 | CC(=O)O | 1.22 |
| sample_4 | C1CCNC1 | 1.15 |
| sample_5 | NC(=O)NO | 1.12 |
3.GNN(Graph Neural Network)、GCN(Graph Convolutional Networks)
3.1 GNN(Graph Neural Network)
グラフニューラルネットワーク(Graph Neural Network, GNN)は、グラフ構造を持つデータに対して効果的な機械学習手法です。グラフはノード(頂点)とエッジ(辺)からなるデータ表現であり、GNNはこれらのデータを入力として受け取り、ノードやエッジ間の相互作用をモデル化します。今回扱う化合物データをノードを原子、エッジを化学結合とみなすとグラフデータに相当します。
GNNでは、各ノードの数値化された特徴表現を用いて、グラフ分類、ノード分類、リンク予測などを解決するための予測や分類が行えます。
今回扱う化合物データの物性予測では1つのグラフ構造(化合物)に1つの値(物性値)が紐づき、それを回帰する問題のためグラフ分類(回帰)問題になります。
GNNの利点の一つは、グラフ構造の情報を効果的に抽出できることです。GNNは、ノードやエッジの局所的な特徴だけでなく、ネットワーク全体の構造や相互作用を次節で示すGCN(Graph Convolutional Networks)で考慮することができます。そのため、グラフデータにおいては、ノード間の依存関係や隣接関係を考慮する必要があるさまざまなタスクにおいて、GNNは優れた性能を発揮することが期待できます。
3.2 GCN(Graph Convolutional Networks)
GNN(Graph Neural Network)では、グラフ畳み込み(Graph Convolution)によりグラフデータの特徴を抽出します。
グラフ畳み込み(Graph Convolution)は、グラフデータに対して畳み込み操作を適用する手法です。通常の畳み込みは、画像などのグリッド状のデータに対して行われますが、グラフ畳み込みは非ユニフォームなグラフ構造に対しても適用することができます。
以下にグラフ畳み込みによる特徴抽出の流れを示します。
- ノードの特徴表現の更新: グラフ畳み込みでは、各ノードの特徴表現を更新するために近傍のノードとの情報を利用します。各ノードは初期的に特徴ベクトルを持ち、周囲のノードとの情報を統合することで新しい特徴表現を計算します。
- 近傍ノードの集約: グラフ畳み込みでは、各ノードの近傍ノード(隣接するノード)との情報を集約します。この集約は、隣接するノードの特徴表現を重み付けして統合することで行われます。一般的な手法として、隣接ノードの特徴を線形結合したり、メッセージパッシングと呼ばれる手法を用いて情報を伝播させる方法があります。
- フィルタリング: グラフ畳み込みでは、集約された情報に対してフィルタリング操作を適用します。フィルタリングは、ノードの特徴表現を調整するために使用されます。一般的には、フィルタリング操作には、パラメータ化された重み行列を使用します。
これらの手順を複数の階層で反復的に行うことで、グラフ畳み込みは特徴表現の階層的な抽象化を実現します。情報は近傍ノードを介して伝播し、隣接関係やグラフの構造を考慮しながら特徴表現が更新されます。
Pytorch Geometricには複数のグラフ畳み込みレイヤーが実装されていますが、今回はGCNConv()レイヤーを使用します。
GCNConvレイヤーは下記の論文の内容が実装されているようです。
https://arxiv.org/pdf/1609.02907.pdf
また、下記の記事では上記の論文で提案されているGCNConvレイヤーについて簡単にまとめられています。
qiita.com
GCNConvレイヤーでは、ノード(原子)の情報とノード同士の隣接関係を使用して畳み込み演算を行い、グラフ構造の特徴を抽出するシンプルなグラフ畳み込みです。
そのほかにも、エッジに重みをつけて畳み込みする(ノードの結合の強さを考慮して学習する)等のGCNレイヤーが提案されており、Pytorch Geometricでも実装されています。
4. 化合物の水溶解度予測モデルの構築
以下の流れで水溶解度予測モデルの学習をしていきます。
- データセットの作成:SMILESデータをMolオブジェクトに変換し、ノード情報及びノードの隣接関係を数値化してPytorch Gemotricで扱える形に変換する。
- GCNN(Graph Convolutional Neural Network)の学習:GCNNを定義し、学習を行う。
4.1 ライブラリのインポート
import numpy as np import pandas as pd from rdkit import Chem import torch from torch_geometric.data import Dataset, Data from torch.nn.functional import one_hot import os import seaborn as sns sns.set()
4.2 データセットの作成
PyTorch Geometricのグラフデータを作成するためのクラスtorch_geometric.data.Datasetを使用してSMILESデータをグラフデータに変換します。
pytorch-geometric.readthedocs.io 上記がPyTorch Geometricが提供しているDatasetクラスのソースコードです。
@property def raw_file_names(self) -> Union[str, List[str], Tuple]: r"""The name of the files in the :obj:`self.raw_dir` folder that must be present in order to skip downloading.""" raise NotImplementedError @property def processed_file_names(self) -> Union[str, List[str], Tuple]: r"""The name of the files in the :obj:`self.processed_dir` folder that must be present in order to skip processing.""" raise NotImplementedError def download(self): r"""Downloads the dataset to the :obj:`self.raw_dir` folder.""" raise NotImplementedError def process(self): r"""Processes the dataset to the :obj:`self.processed_dir` folder.""" raise NotImplementedError @abstractmethod def len(self) -> int: r"""Returns the number of graphs stored in the dataset.""" raise NotImplementedError @abstractmethod def get(self, idx: int) -> BaseData: r"""Gets the data object at index :obj:`idx`.""" raise NotImplementedError
このあたりの関数には、何も定義されていませんので、今回扱う問題に対応できるようにオーバーライドしてカスタマイズします。
class MoleculeDataset(Dataset): def __init__(self, root, filename, test=False, transform=None, pre_transform=None): #データを読み込むファイル名を指定する。 self.filename = filename super(MoleculeDataset, self).__init__(root, transform, pre_transform) @property def raw_file_names(self): return self.filename @property def processed_file_names(self): self.data = pd.read_csv(self.raw_paths[0], index_col=0).reset_index() #グラフデータに変換したものをバイナリ化して格納するファイル名を指定する。 return [f'data_{i}.pt' for i in list(self.data.index)] def download(self): pass def len(self): return self.data.shape[0] #インスタンス化した後に各グラフデータ(化合物)を呼び出す際に使用する。 #インスタンス[idx]の形式で各グラフデータを呼び出せる。(特殊メソッド__getitem__の中にgeメソッドがあるため)) def get(self, idx): idx = idx + 1 data = torch.load(os.path.join(self.processed_dir, f'data_sample_{idx}.pt')) return data #分子グラフを作成する関数を記述する。 def process(self): #データをcsvファイルから読み込む。 self.data = pd.read_csv(self.raw_paths[0], index_col=0) #一つずつSMILESデータを読み込んで、Molオブジェクトに変換し、ノード情報及び隣接関係を数値化する。 for index, mol in self.data.iterrows(): mol_obj = Chem.MolFromSmiles(mol[0]) #ノード(原子)情報を数値化 node_feats = self._get_node_features(mol_obj) #エッジ(化学結合)の情報を数値化 edge_feats = self._get_edge_features(mol_obj) #原子の隣接関係を数値化 edge_index = self._get_adjacency_info(mol_obj) #教師ラベル label = self._get_labels(mol[1]) structure_id = [[mol[0]]] #Dataクラスをインスタン化してノード情報、エッジ情報、隣接関係をまとめてグラフデータ化する。 data = Data(x=node_feats, edge_index=edge_index, edge_attr=edge_feats, y=label, structure_id=structure_id) #グラフデータを保存する。 torch.save(data, os.path.join(self.processed_dir, f'data_{index}.pt'))
processメソッドの中でcsvファイル(./dataset/raw/に配置)を読み込んでノード情報、エッジ情報、隣接関係を数値化して化合物ごとにデータを作成し、バイナリ化してデータを格納します。 processメソッドはインスタンス化する際に呼び出され、インスタンス化した時点でグラフデータが揃います。 また、processメソッド内で使用するノード情報量等を算出する関数を同一クラス内で定義しておきます。 ノード情報量や隣接関係等はrdkitを使用します。
def _get_node_features(self, mol): all_node_feats = [] for atom in mol.GetAtoms(): node_feats = [] node_feats = one_hot(torch.tensor(atom.GetAtomicNum()-1), num_classes=113) node_feats = node_feats.tolist() node_feats.append(atom.GetDegree()) node_feats.append(atom.GetFormalCharge()) node_feats.append(atom.GetHybridization()) node_feats.append(atom.GetIsAromatic()) node_feats.append(atom.GetTotalNumHs()) node_feats.append(atom.GetNumRadicalElectrons()) node_feats.append(atom.IsInRing()) node_feats.append(atom.GetChiralTag()) all_node_feats.append(node_feats) all_node_feats = np.asarray(all_node_feats) return torch.tensor(all_node_feats, dtype=torch.float) def _get_edge_features(self, mol): all_edge_feats = [] for bond in mol.GetBonds(): edge_feats = [] edge_feats.append(torch.tensor(bond.GetBondTypeAsDouble())) edge_feats.append(bond.IsInRing()) all_edge_feats += [edge_feats, edge_feats] all_edge_feats = np.asarray(all_edge_feats) return torch.tensor(all_edge_feats, dtype=torch.float) def _get_adjacency_info(self, mol): edge_indices = [] for bond in mol.GetBonds(): i = bond.GetBeginAtomIdx() j = bond.GetEndAtomIdx() edge_indices += [[i, j], [j,i]] edge_indices = torch.tensor(edge_indices) edge_indices = edge_indices.t().to(torch.long).view(2, -1) return edge_indices def _get_labels(self, label): label = np.asarray([label]) label = torch.tensor(label, dtype=torch.float32) return torch.reshape(label, (1,1))
ここまででデータセット作成クラスの定義は完了です。以下のコードでデータセットをインスタンス化します。
dataset = MoleculeDataset(root='./dataset/', filename="molecules.csv")
下記のようにコンソールに表示されれば完了です。
Processing... Done!
また、下記のようにインスタンスに対して
dataset[0]
とすると、インスタンス内の[]で指定したインデックスの化合物データのグラフデータが得られます。
Data(x=[4, 121], edge_index=[2, 6], edge_attr=[6, 2], y=[1, 1], structure_id=[1])
4.3 学習用データへの変換
from torch_geometric.loader import DataLoader dataset = dataset.shuffle() #学習データとテストデータに分割する。 from sklearn.model_selection import train_test_split dataset_train, dataset_test = train_test_split(dataset, test_size=0.2) #バッチサイズ batch_size = 64 loader_train = DataLoader(dataset_train, batch_size=batch_size, shuffle=True) loader_test = DataLoader(dataset_test, batch_size=batch_size, shuffle=False)
4.4 GCNN(Graph Convolutional Neural Network)の定義
2層のグラフ畳み込み層を設定しました。畳み込みの後はglobal_mean_poolで固定長に変化し、Affine繋げます。
import torch import torch.nn as nn from torch_geometric.nn import GCNConv from torch_geometric.nn import global_mean_pool #ノードの隠れ層の数 n_h = 64 class GCN(nn.Module): def __init__(self): super().__init__() self.conv1 = GCNConv(dataset.num_node_features, n_h) self.conv2 = GCNConv(n_h, n_h) self.conv3 = GCNConv(n_h, n_h) self.fc1 = nn.Linear(n_h, n_h//2) self.fc2 = nn.Linear(n_h//2, 1) self.relu = nn.ReLU() self.dropout = nn.Dropout(p=0.5) def forward(self, data): x = data.x edge_index = data.edge_index batch = data.batch x = self.conv1(x, edge_index) x = self.relu(x) x = self.conv2(x, edge_index) x = self.relu(x) x = self.conv3(x, edge_index) x = global_mean_pool(x, batch) x = self.dropout(x) x = self.fc1(x) x = self.relu(x) x = self.fc2(x) return x #ネットワークのインスタンス化 net = GCN()
4.5 GCNN(Graph Convolutional Neural Network)の学習
100epochの学習を行い、その際のロスのデータを保存します。
from torch import optim def eval(loader, loss_fn): for data in loader: out = net(data) loss = loss_fn(out, data.y) return loss loss_fn = nn.L1Loss() record_loss_test = [] record_loss_train = [] optimizer = optim.AdamW(net.parameters()) for epoch in range(100): #学習モード net.train() for data in loader_train: optimizer.zero_grad() out = net(data) loss = loss_fn(out, data.y) loss.backward() optimizer.step() #評価モード net.eval() loss_train = eval(loader_train, loss_fn) loss_test = eval(loader_test, loss_fn) record_loss_train.append(loss_train.item()) record_loss_test.append(loss_test.item()) if (epoch+1)%10==0: print('Epoch:', epoch+1, "MAE_train:", loss_train.item(), "MAE_test:", loss_test.item())
学習完了後、学習曲線を描画します。
import matplotlib.pyplot as plt plt.plot(range(len(record_loss_train)), record_loss_train, label="Train") plt.plot(range(len(record_loss_test)), record_loss_test, label="Test") plt.legend() plt.xlabel('Epochs') plt.ylabel('Loss') plt.show()

4.6 予測精度の確認
バリデーションデータに対する予測を行い、実測値と予測値をプロットして精度を確認してみます。
net.eval() # ネットワークを推論モードに切り替える predictions = [] labels = [] with torch.no_grad(): for data in loader_test: label = data.y output = net(data) # ネットワークにテストデータを入力して予測結果を取得 labels.append(label) predictions.append(output) predictions = torch.cat(predictions, dim=0) # 予測結果を結合して1つのテンソルにする labels = torch.cat(labels, dim=0) plt.scatter(labels.float(), predictions.float()) plt.xlabel('Measured') plt.ylabel('Predicted') plt.show()
 それなりの精度で予測できていそうです。
それなりの精度で予測できていそうです。
5 最後に
今回、GCN(Graph Convolutional Networks)を使用して化合物データの水溶解予測をしてみました。 そのほかのGNNも色々調べて、実装していきたいと思います。 今期からMI(マテリアルインフォマティクス)テーマに参画させてもらっているので、MI関連技術調査に力を入れていきたいです。
Flask+Vue.jsでベイズ最適化アプリを作成してみた
0.はじめに
Flask + Vue.jsで簡単なベイズ最適化アプリを作成したので備忘録がてらまとめておきます。
1.つくりたいもの
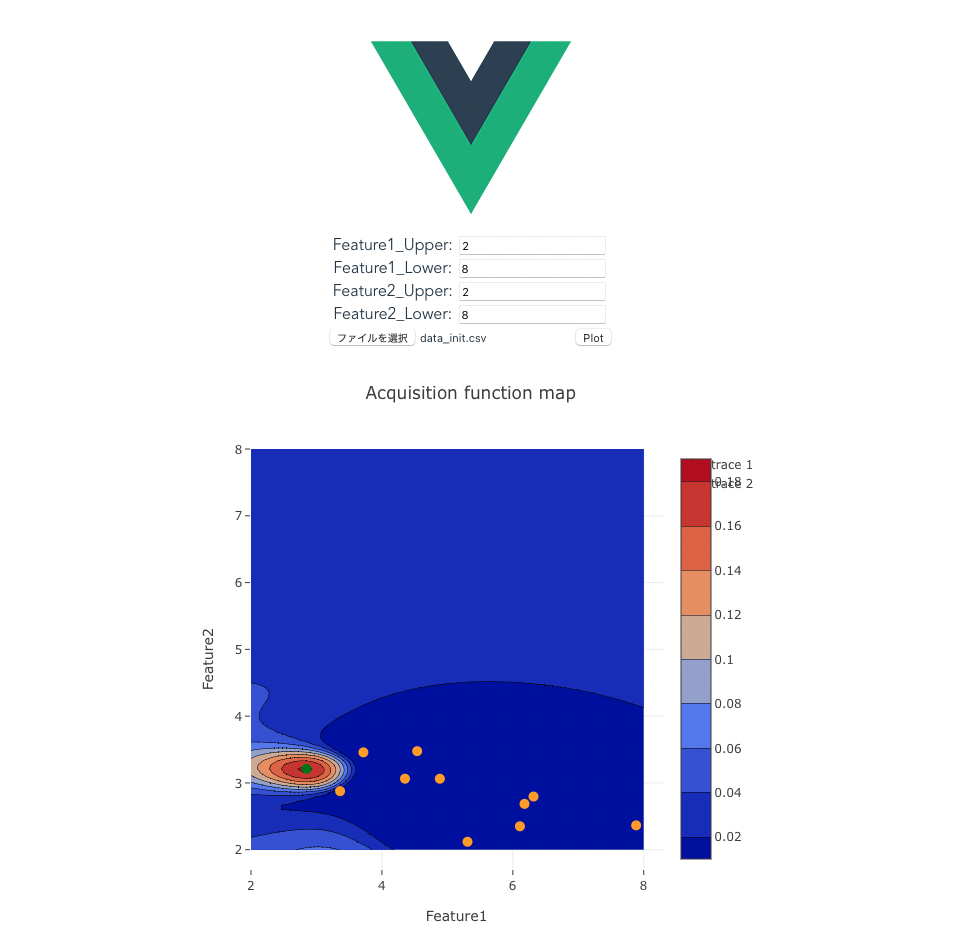
以下のような、csv形式のテーブルデータ(property:目的変数、feature:説明変数、説明変数は2つ)を読み込んで目的変数を最大化する条件探索をベイズ最適化によって行うwebアプリケーションを作成します。
vue.jsで作成したフロントエンドで各変数の取りうる上下限値をフォームに入力、探索初期サンプルの入出力が記載されたcsv形式のデータを読み込みPlotボタンを押すとバックエンドで読み込んだデータを使用してベイズ最適化が行われ、獲得関数マップが表示されます。
また、獲得関数をもとに次の探索候補点(緑点)が表示されます。実験者は次の探索候補点の条件で再度データ取得を行い、取得したデータを追加し、再度アプリケーションを使用することで対話的に目的変数を最大化する条件の最適化が行えます。
初期サンプルデータ
| No | property | feature_1 | feature_2 |
|---|---|---|---|
| sample_1 | -1.6901378073208400 | 6.17881511358717 | 2.686356032301740 |
| sample_2 | -0.19462331210335700 | 3.716836009702280 | 3.4580994147680800 |
| sample_3 | -0.05709174688868170 | 3.361108721385220 | 2.8771444893592500 |
| sample_4 | -0.5455318144995780 | 5.307888614497350 | 2.1193557932191400 |
| sample_5 | -1.8222159386202400 | 6.316813818713380 | 2.796088510660860 |
作成するアプリの出力
2.バックエンド
バックエンドではフロントエンドで入力したデータと説明変数の上下値を受け取り、ベイズ最適化を行います。
ベイズ最適化によって得られた獲得関数、次の探索候補点座標をjson形式に変換し、フロントエンドへ渡します。
app.py内に定義したエンドポイントをフロントから呼び出すことで上記のjson形式データを渡します。
ベイズ最適化関数はgpbo.py内に関数を定義します。
app.pyとgpbo.pyは同じ階層に配置しておきます。
2.1 事前準備
以下のライブラリをインストールしておきます。
pip install flask pip install flask_cors pip install pandas pip install numpy pip install scipy pip install scikit-learn
2.2 スクリプト
app.py
from flask import Flask, jsonify, request, send_file from flask_cors import CORS import pandas as pd import numpy as np from gpbo import gpbo app = Flask(__name__) cors = CORS(app, resources={r"/api/*": {"origins":"*"}} ) #エンドポイントの作成 @app.route('/api/v1.0/gpbo', methods=['POST']) def upload(): # フロントエンドで入力したCSVファイルを受け取る csv_file = request.files['file'] # ファイルをPandasのデータフレーム形式に変換する df = pd.read_csv(csv_file, index_col=0) print(df.head()) print('ファイルをアップロードしました') #探索の上下限の設定 lower_feature1 = int(request.form['Feature1_Lower']) upper_feature1 = int(request.form['Feature1_Upper']) lower_feature2 = int(request.form['Feature2_Lower']) upper_feature2 = int(request.form['Feature2_Upper']) # x, y の値域を指定(探索範囲内メッシュデータ、獲得関数マップを描画する目的) x_= np.linspace(lower_feature1, upper_feature1, 100) y_ = np.linspace(lower_feature2, upper_feature2, 100) X, Y = np.meshgrid(x_, y_) #ベイズ最適化により次の探索点を提案、獲得関数の値 z_, next_sample = gpbo(df, X, Y) #これまでのサンプルデータ、探索範囲内データ、次の探索点をjson形式で出力する。 data = {'x':df.iloc[:,1].values.tolist(), 'y':df.iloc[:,2].values.tolist(),'x_':x_.tolist(), 'y_':y_.tolist(), 'z_':z_.tolist(),'x_next':next_sample.iloc[:,0].values.tolist(), 'y_next':next_sample.iloc[:,1].values.tolist()} return jsonify(data) if __name__ == '__main__': app.run(debug=True)
gpbo.py
import pandas as pd import numpy as np from scipy.stats import norm from sklearn.model_selection import KFold, cross_val_predict from sklearn.gaussian_process import GaussianProcessRegressor from sklearn.gaussian_process.kernels import WhiteKernel, RBF, ConstantKernel, Matern, DotProduct from sklearn.metrics import r2_score, mean_squared_error, mean_absolute_error def gpbo(dataset, X, Y): acquisition_function = 'EI' fold_number = 4 # クロスバリデーションの fold 数 kernel_number = 2 # 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 relaxation = 0.01 # EI # データ分割 y = dataset.iloc[:, 0] # 目的変数 x = dataset.iloc[:, 1:] # 説明変数 #テストデータの設定 X_ = X.reshape(-1) Y_ = Y.reshape(-1) x_prediction = pd.DataFrame() x_prediction['feature_1'] = X_ x_prediction['feature_2'] = Y_ autoscaled_x_prediction = (x_prediction - x.mean()) / x.std() # カーネル 11 種類 kernels = [ConstantKernel() * DotProduct() + WhiteKernel(), ConstantKernel() * RBF() + WhiteKernel(), ConstantKernel() * RBF() + WhiteKernel() + ConstantKernel() * DotProduct(), ConstantKernel() * RBF(np.ones(x.shape[1])) + WhiteKernel(), ConstantKernel() * RBF(np.ones(x.shape[1])) + WhiteKernel() + ConstantKernel() * DotProduct(), ConstantKernel() * Matern(nu=1.5) + WhiteKernel(), ConstantKernel() * Matern(nu=1.5) + WhiteKernel() + ConstantKernel() * DotProduct(), ConstantKernel() * Matern(nu=0.5) + WhiteKernel(), ConstantKernel() * Matern(nu=0.5) + WhiteKernel() + ConstantKernel() * DotProduct(), ConstantKernel() * Matern(nu=2.5) + WhiteKernel(), ConstantKernel() * Matern(nu=2.5) + WhiteKernel() + ConstantKernel() * DotProduct()] # オートスケーリング autoscaled_y = (y - y.mean()) / y.std() autoscaled_x = (x - x.mean()) / x.std() autoscaled_x_prediction = (x_prediction - x.mean()) / x.std() # モデル構築 selected_kernel = kernels[kernel_number] model = GaussianProcessRegressor(alpha=0, kernel=selected_kernel) model.fit(autoscaled_x, autoscaled_y) # モデル構築 # 予測 estimated_y_prediction, estimated_y_prediction_std = model.predict(autoscaled_x_prediction, return_std=True) estimated_y_prediction = estimated_y_prediction * y.std() + y.mean() estimated_y_prediction_std = estimated_y_prediction_std * y.std() # 獲得関数の計算 acquisition_function_prediction = (estimated_y_prediction - max(y) - relaxation * y.std()) * norm.cdf((estimated_y_prediction - max(y) - relaxation * y.std()) / estimated_y_prediction_std) + estimated_y_prediction_std * norm.pdf((estimated_y_prediction - max(y) - relaxation * y.std()) / estimated_y_prediction_std) acquisition_function_prediction[estimated_y_prediction_std <= 0] = 0 estimated_y_prediction = pd.DataFrame(estimated_y_prediction, x_prediction.index, columns=['estimated_y']) estimated_y_prediction_std = pd.DataFrame(estimated_y_prediction_std, x_prediction.index, columns=['std_of_estimated_y']) acquisition_function_prediction = pd.DataFrame(acquisition_function_prediction, index=x_prediction.index, columns=['acquisition_function']) # 次のサンプル next_sample = x_prediction.loc[acquisition_function_prediction.idxmax()] return acquisition_function_prediction.values.reshape(X.shape[0], Y.shape[0]), next_sample
2.3 実行
以下のコマンドを実行して、バックエンドサーバーを立ち上げます。
python app.py
すると、http://localhost:5000にバックエンドサーバーが立ち上がります。
3.フロントエンド
Vue.js(Vue-CLI)を使用してフロントエンドを作成します。 csvデータとフォームに入力した説明変数の上下値をバックエンドに渡し、バックエンドでベイズ最適化が行われた戻り値を受け取り、コンター図を描画する機能を実装します。
3.1 事前準備
node.jsをインストールしておく必要があります。node.jsのインストール
インストール後、npmコマンドが実行できることを確認してください。
以下のコマンドを実行してVueをインストールします。
npm install -g @vue/cli
vueの新しいプロジェクトを作成します。
npm init webpack my-project
(Y / n)を押して選択
矢印キーで選択してEnter
? Project name my-project ? Project description A Vue.js project ? Author ? Vue build standalone ? Install vue-router? Yes ? Use ESLint to lint your code? Yes ? Pick an ESLint preset Standard ? Set up unit tests Yes ? Pick a test runner jest ? Setup e2e tests with Nightwatch? No ? Should we run `npm install` for you after the project has been created? (recommended) npm
質問が終わるとプロジェクトの作成が始まります。
終了すると、実行ディレクトリ/プロジェクト名配下にファイルが作成されました。
ファイルが作成されたことを確認し、
cd my-project npm run dev
上記のコマンドを実行すると、vueアプリケーションがhttp://localhost:8080に立ち上がります。
アクセスして確認してください。
今回のアプロケーションではflaskバックエンドapiとhttpクライアントのaxiosによって通信を行うので、axiosをインストールします。
npm install axios --save
また、flaskバックエンドapiで受け取ったデータを用いてフロントエンドで獲得関数マップの描画を行うため、plotly.jsをインストールします。
npm install plotly.js
3.2 ディレクトリ構造
3.1でvueプロジェクトを作成すると以下のディレクトリが作成されています。
. ├── README.md ├── build ├── config ├── index.html ├── node_modules ├── package-lock.json ├── package.json ├── src └── static
今回は./srcディレクトリ内のcomponentsディレクトリ内にFigure.vueを追加し、Figure.vueをflaskバックエンドapiを受け取って、獲得関数マップを表示するフロントエンドして機能させます。
また、index.jsにFigure.vueのルーティングを追加します。
.
├── App.vue
├── assets
│ └── logo.png
├── components
│ ├── Figure.vue
│ ├── HelloWorld.vue
|
├── main.js
└── router
└── index.js
3.3 スクリプト
Figure.vue
<template> <div> <form> <div> <label for="feature1_upper">Feature1_Upper:</label> <input type="text" id="feature1_upper" v-model="feature1_upper"> </div> <div> <label for="feature1_lower">Feature1_Lower:</label> <input type="text" id="feature1_lower" v-model="feature1_lower"> </div> <div> <label for="feature2_upper">Feature2_Upper:</label> <input type="text" id="feature2_upper" v-model="feature2_upper"> </div> <div> <label for="feature2_lower">Feature2_Lower:</label> <input type="text" id="feature2_lower" v-model="feature2_lower"> </div> </form> <input type="file" ref="fileInput" @change="onFileSelected"> <button @click="drawPlot" :disabled="!dataLoaded">Plot</button> <div ref="plot" class="plot"></div> </div> </template> <script> import axios from 'axios' import Plotly from 'plotly.js' export default { data () { return { feature1_upper: '', feature1_lower: '', feature2_upper: '', feature2_lower: '', x_: [], y_: [], z_: [], x: [], y: [], x_next: [], y_next: [], dataLoaded: false } }, methods: { onFileSelected (event) { const file = event.target.files[0] const formData = new FormData() formData.append('file', file) formData.append('Feature1_Upper', this.feature1_upper) formData.append('Feature1_Lower', this.feature1_lower) formData.append('Feature2_Upper', this.feature2_upper) formData.append('Feature2_Lower', this.feature2_lower) axios.post('http://127.0.0.1:5000/api/v1.0/gpbo', formData) .then(response => { this.x = response.data.x this.y = response.data.y this.x_ = response.data.x_ this.y_ = response.data.y_ this.z_ = response.data.z_ this.x_next = response.data.x_next this.y_next = response.data.y_next this.dataLoaded = true }) .catch(error => { console.log(error) }) }, drawPlot () { const trace = { x: this.x_, y: this.y_, z: this.z_, type: 'contour' } const scatterTrace = { x: this.x, y: this.y, mode: 'markers', type: 'scatter', marker: { color: 'orange', // 色をzに設定する size: 10 // サイズを10に設定する } } const scatterTrace2 = { x: this.x_next, y: this.y_next, mode: 'markers', type: 'scatter', marker: { color: 'green', // 色をzに設定する size: 10 // サイズを10に設定する } } const layout = { title: 'Acquisition function map', xaxis: { title: 'Feature1' }, yaxis: { title: 'Feature2' } } Plotly.newPlot(this.$refs.plot, [trace, scatterTrace, scatterTrace2], layout) } } } </script> <style> .plot { width: 600px; height: 600px; margin: auto; } </style>
※バックエンドのローカルアドレスをhttp://localhost:5000とするとエラーが出てしまいました。
index.js
import Vue from 'vue' import Router from 'vue-router' import Figure from '@/components/Figure' Vue.use(Router) export default new Router({ mode: 'history', routes: [ { path: '/figure', name: 'Figure', component: Figure } ] })
3.4 実行
npm run dev
を実行するとフロントエンドサーバーがhttp://localhost:8080に立ち上がります。
今回作成したベイズ最適化アプリはhttp://localhost:8080/figureに立ち上がります。

4.使用例
下記のような解が既知である問題に対して、今回作成したベイズ最適化アプリを使って目的変数が最大となる解の探索を行なってみます。

出力関数は以下です。
def f(x,y): return np.cos(x) + np.cos(y)+ np.cos(x+y)+ np.cos(x-y)
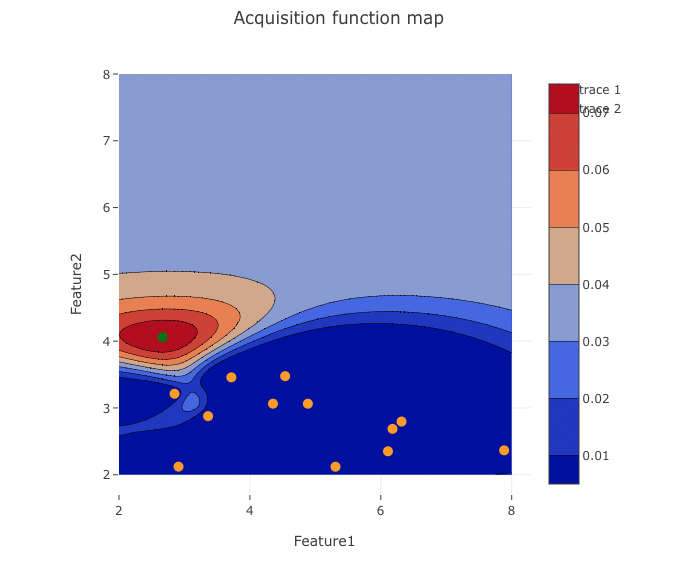
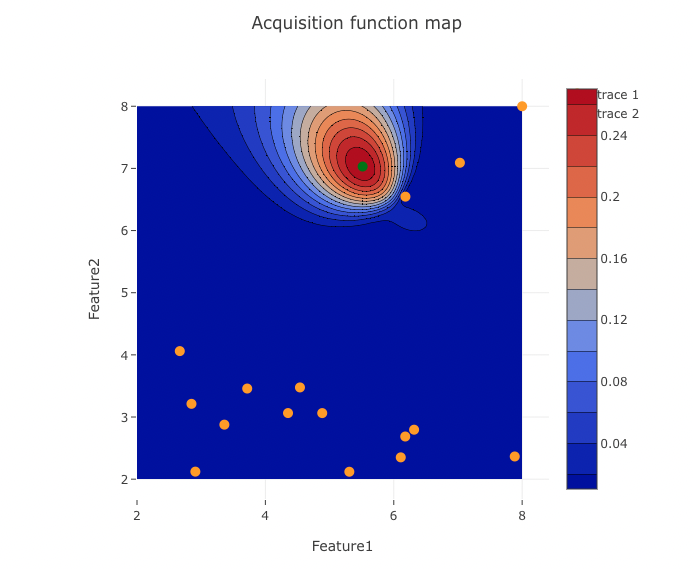
初期サンプル(オレンジの点)を10点とり、ベイズ最適化を実行し、獲得関数をアプリで可視化、次の探索点で評価(上式に代入)、テーブルデータに追加を繰り返します。
1回目
2回目
 3回目
3回目
4回目

作成したベイズ最適化アプリを使用することで獲得関数マップを確認しつつ、8回の試行でおおよそ目的変数が最大となる2変数の組み合わせを見つけられました。
5.参考
https://github.com/hkaneko1985/python_doe_kspub
Vue.js(vue-cli)とFlaskを使って簡易アプリを作成する【前半 - フロントエンド編】
Vue.js(vue-cli)とFlaskを使って簡易アプリを作成する【前半 - バックエンド編】
Flaskを勉強するのに下記の書籍を参考にしました。
PLS(部分的最小二乗法)の変数重要度をvip法を使って算出してみた
1. 背景
化学工学における物性値の予測やスペクトル解析においては、サンプル数に対して説明変数(特徴量)が非常に大きいということがある。そのような場合、一般的なOLS(最小二乗法)等の線形回帰モデルが使用できない。また、説明変数同士の相関が大きい(多重共線性、マルチコ)、適切な回帰モデルが構築できないという問題がある。このような問題に対して、PLS(部分的最小二乗法)が有効な手法である。PLSでは説明変数同士の相関をPCAにより排除し、マルチコを回避できる。また、PCAによりサンプル数が説明変数に比べ小さい場合でも回帰モデルが構築できる。また、目的変数との相関も考慮しながら、モデル が構築されるため、精度も期待できる。PLSはスモールデータ解析においてよくみられる手法の一つである。また、データ解析時には、説明変数の重要度が示せると、モデルの解釈ができたり、重要度の高い変数だけを残して、再度、モデルを学習させることで、汎化性能を高めることが可能である場合がある。PLSにはPLS専用の変数重要度算出方法(PLS-vip法)が提案されている。今回は、PLS-vip法を実装してみたいと思う。
2. PLS(部分的最小二乗法)
手法概要
PLSの導出、PLS-vip法の詳細な説明は他に譲ります。ここでは簡単な説明だけさせて頂きます。
- 部分最小二乗法 (Partial Least Squares, PLS) は、複数の互いに関連する説明変数 (predictor variables) がある場合に、それらの説明変数を少数の線形結合によって表現することで、目的変数 (response variable) の予測を行う回帰分析手法です。
- PLSは、主成分分析 (PCA) と回帰分析を組み合わせた手法であり、まず最初に説明変数の共分散行列を分解し、主成分を抽出します。次に、目的変数との共分散が大きい主成分を抽出し、それを基に部分的な回帰係数を求めます。この回帰係数を使って説明変数を線形結合することで、目的変数の予測を行います。
- PLSは、説明変数が多いデータセットや、説明変数同士の相関が高い場合に有用です。また、変数選択や特徴量抽出を行わずに、説明変数の情報を最大限に利用できるため、データ解析の精度を向上させることができます。
PLS-vip法(特徴量重要度)
- PLS-VIP (Variable Importance in Projection)法は、部分最小二乗法 (PLS)を用いた回帰分析において、各説明変数が目的変数に与える影響度を評価する方法の一つです。
- PLS-VIP法では、各説明変数の重要度を、それぞれの説明変数がPLSの射影に与える寄与度によって評価します。すなわち、PLSの射影において、各説明変数がどの程度目的変数との関連性があるかを測定し、その寄与度を基に、説明変数の重要度を算出します。重要度が高い説明変数ほど、目的変数に対する影響度が大きいと判断されます。
3. やりたいこと
こちらの水溶解度データセット(1290条件のSMILESデータとそれに対応する水溶解度logSを使用して、記述子から水溶解度logSを回帰するPLSモデルを構築し、PLS-vip法によって特徴量重要度を算出すること。
4. PLS-vip法により水溶解度予測モデルにおける各記述子の重要度を求めてみる。
4.1 データの準備
使用するデータを落としてきます。
#水溶解度データセット(SMILES)をスクレイピング
import urllib.request # URLで指定したファイルをダウンロードするライブラリ
url = 'http://modem.ucsd.edu/adme/data/databases/logS/data_set.dat'
urllib.request.urlretrieve(url, 'water_solubility.txt') データを整形します。
import pandas as pd
dataset = pd.read_csv('water_solubility.txt', sep='\t', header=None) # データの読み込み
dataset = dataset.drop([1],axis=1)
dataset.columns = ['SMILES', 'logS']
dataset.head()
rdkitを使用して、SMILESデータを記述子に変換します。
import pandas as pd
from rdkit import Chem
from rdkit.Chem import Descriptors
from rdkit.ML.Descriptors import MoleculeDescriptors
# 計算する記述子名の取得
descriptor_names = []
for descriptor_information in Descriptors.descList:
descriptor_names.append(descriptor_information[0])
print('計算する記述子の数 :', len(descriptor_names))
#記述子の計算
descriptor_calculator = MoleculeDescriptors.MolecularDescriptorCalculator(descriptor_names)
descriptors = [] # ここに計算された記述子の値を追加
for index, smiles_i in enumerate(smiles):
print(index + 1, '/', len(smiles))
molecule = Chem.MolFromSmiles(smiles_i)
descriptors.append(descriptor_calculator.CalcDescriptors(molecule))
descriptors = pd.DataFrame(descriptors, index=dataset.index, columns=descriptor_names)
if dataset.shape[1] > 1:
descriptors = pd.concat([y, descriptors], axis=1) # y と記述子を結合
#欠損値を含むデータがあるので削除
descriptors = descriptors.dropna(how='any')
学習データとバリデーションデータに分割します。
#説明変数と目的変数に分ける
X = descriptors.iloc[:,1:]
y = descriptors.iloc[:, 0]
#標準偏差が0の記述子を削除する
del_descriptors = X.columns[X.std() == 0]
X = X.drop(del_descriptors, axis=1)
#オートスケーリング
X_scaled = (X - X.mean()) / X.std()
y_scaled = (y - y.mean()) / y.std()
4.2 PLSモデルの構築
今回、PLSモデルのハイパーパラメーターである説明変数Xの潜在空間上でのサイズ(n_component)は54に設定した。
from sklearn.model_selection import train_test_split
from sklearn.cross_decomposition import PLSRegression
from sklearn.metrics import r2_score, mean_squared_error
import numpy as np
#学習データとテストデータに分割する
X_train, X_val, y_train, y_val = train_test_split(X_scaled, y_scaled, test_size=0.2, random_state=5, shuffle=True)
#plsの圧縮サイズを指定する
n_component = 54
pls = PLSRegression(n_components=n_component)
pls.fit(X_train, y_train.values.reshape(-1))
pred_train = pls.predict(X_train)
pred_val = pls.predict(X_val)
print('R2_score_train', r2_score(pred_train, y_train))
print('R2_score_val', r2_score(pred_val, y_val))
print('RMSE_train', np.sqrt(mean_squared_error(pred_train, y_train)))
print('RMSE_val', np.sqrt(mean_squared_error(pred_val, y_val)))4.3 特徴量重要度の可視化(PLS-vip)
続いて以下のスクリプトでpls-vip値を算出する関数を定義する。scikit-learnでplsモデルを学習させた後に得られる重み行列(x_weights_),目的変数yに対するローディング行列y_loadings_及び説明変数Xの潜在空間での値(x_scores_)を用いて算出できる。
#PLS-vop値を算出する関数
def pls_vip(pls_model):
W = pls_model.x_weights_
D = pls_model.y_loadings_
T = pls_model.x_scores_
M, R = W.shape
weight = np.zeros([R])
vips = np.zeros([M])
ssy = np.diag(T.T @ T @ D.T @ D)
total_ssy = np.sum(ssy)
for m in range(M):
for r in range(R):
weight[r] = np.array([(W[m, r]/ np.linalg.norm(W[:, r]))**2])
vips[m] = np.sqrt(M*(ssy @ weight) / total_ssy)
sel_var = np.arange(M)
#vip値が1よりも大きい特徴量名前を抽出する
sel_var = sel_var[vips>=1]
return sel_var, vips
上記の関数を使用してpls-vip値を算出して特徴量重要度を値が大きい順に並べて可視化します。
sel_var , vips = pls_vip(pls)
#データフレーム化
vips_df = pd.DataFrame(vips, index=X.columns, columns=['vips'])
vips_df = vips_df.sort_values('vips', ascending=True)
#可視化
import seaborn as sns
sns.set()
plt.figure(figsize=(20, 40))
plt.barh(vips_df.index, vips_df['vips'], align='center')
plt.yticks(fontsize=14)
plt.xticks(fontsize=14)
plt.title('Feature importance (PLS-vip)',fontsize=20)
plt.show()
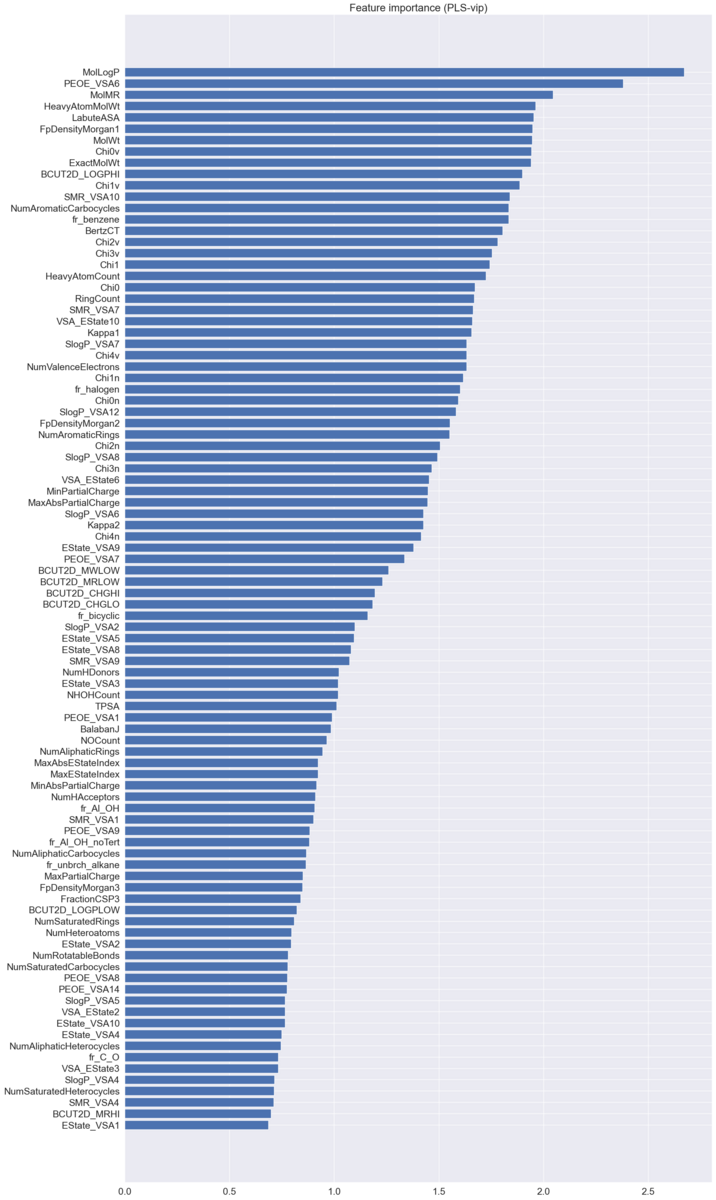
ちなみに、最も重要度の高いMolLogPは水/オクタノール分配係数(水溶性・脂溶性の指標)なので、水溶解度への寄与が大きいことは妥当な結果であると考えられます。
参考
こちらの本でPLSの詳細な導出やpls-vip法の説明が書かれています。参考にしてみてください。
Kedro + MLFlowでMLOpsを導入してみた
1. 背景
データフローパイプラインとモデルの実験管理を行えるようなMLOpsパイプラインを作成したいと思い、調べているとKedro+MLflowでできることがわかりました。実際にKedroとMLFlowを使ってパイプラインを作って実験管理をしてみたので共有します。
2. Kedro
Kedroとは?

Kedroは、Pythonでのデータパイプライン開発を容易にするために設計されたオープンソースのフレームワークです。Kedroを使用することで、データサイエンティストやエンジニアは、高品質のデータパイプラインを迅速に構築できます。
Kedroの特徴
- データセットの抽象化:Kedroは、さまざまな種類のデータソース(CSV、JSON、データベースなど)に対応するために、データセットと呼ばれる抽象化レイヤーを提供しています。これにより、データセットを簡単に交換し、再利用することができます。
- パイプラインの可視化:Kedroは、パイプラインの可視化機能を提供しています。これにより、データの流れを理解しやすくなり、パイプラインのデバッグや最適化が容易になります。
- テストの自動化:Kedroは、pytestと統合されており、自動化されたユニットテストと統合テストを実行することができます。これにより、コンポーネントやパイプラインが正常に動作することを確認できます。
- モジュール性:Kedroは、コンポーネントを再利用可能な形で開発することを容易にします。これにより、コンポーネントの再利用や、パイプラインのカスタマイズが簡単になります。
Kedro公式ページ:
3. MLFlow
MLflowとは?
MLflowは、機械学習の開発ライフサイクルにおいて発生する問題を解決するために作られたプラットフォームです。機械学習の開発ライフサイクルは、データの前処理や特徴量の選択、モデルのトレーニング、モデルのデプロイなど、多岐にわたります。このような開発ライフサイクルにおいては、モデルの開発や実行に関する様々な情報を追跡し、管理する必要があります。MLflowは、このような情報を効率的に管理するためのプラットフォームであり、以下のような機能を提供します。
MLflowの機能
- トラッキング:モデルのトレーニングにおける実行結果やメトリック、パラメータ、入出力をトラッキングするための機能です。
- プロジェクト:モデルのコードや依存ライブラリを管理するための機能です。
- モデルのバージョニング:モデルのバージョン管理を行うための機能です。
- モデルのデプロイ:モデルのデプロイに関する機能です。
MLflowの使い方
MLflowを使用するためには、以下の手順を実行します。
- MLflowをインストールする。
- MLflowトラッキングサーバーを起動する。
- MLflowのAPIを使用して、トラッキング情報を保存する。
- 保存したトラッキング情報を、MLflow UIを使用して閲覧する。
MLFlow公式ページ:
4. ライブラリ
kedroを使用する際は、gitをインストールをしておく必要がある。
以下が使用したライブラリのバージョンです。
python == 3.8.16
numpy == 1.23.5
pandas == 1.5.3
scikit-learn == 1.2.1
kedro == 0.18.5
mlflow == 2.1.1
5. Kedro + MLFlowでパイプライン作成、実験管理をおこなってみる
やりたいこと
ボストンデータセットを使用し、PLS(部分最小二乗法)を使って前処理〜学習までのデータフローパイプラインを作成する。パイプラインのなかでPLSモデルのハイパーパラメータを振って学習させ、その結果をMLFLowに保存して管理する。
データ準備
あらかじめ、以下のスクリプトを実行しデータセットを準備しておく。
import pandas as pd
california_housing = fetch_california_housing()
train_x = pd.DataFrame(california_housing.data, columns=california_housing.feature_names)
train_y = pd.DataFrame(california_housing.target, columns=california_housing.target_names)
raw_df = pd.concat([train_x, train_y], axis=1)
raw_df.to_csv('boston_house_prices.csv')
データフローパイプラインの作成
続いて、以下のコマンドを実行し、kedroパイプラインの雛形を作成します。名前をきかれるのでboston-plsとします。
kedro new
実行すると、以下のディレクトリが作成されます。
./boston-pls/
├── README.md
├── conf
│ ├── README.md
│ ├── base
│ └── local
├── data
│ ├── 01_raw
│ ├── 02_intermediate
│ ├── 03_primary
│ ├── 04_feature
│ ├── 05_model_input
│ ├── 06_models
│ ├── 07_model_output
│ └── 08_reporting
├── docs
│ └── source
├── logs
│ ├── errors.log
│ └── info.log
├── notebooks
├── pyproject.toml
├── setup.cfg
└── src
├── boston_pls
├── requirements.txt
├── setup.py
└── tests
続いて、あらかじめ出力しておいた今回使用するcsvファイルを./data/01_rawに格納し、./conf/base/catalog.ymlに以下のスクリプトを追加することでcsvファイルの読み込みがパイプラインに組み込まれます。
boston:
type: pandas.CSVDataSet
filepath: data/01_raw/boston_house_prices.csv
これにより、bostonという名前のデータセットが定義され、PandasのCSVDataSetを使用して、data/01_raw/boston_house_prices.csvファイルを読み込むことができます。
次に以下のコマンドを実行し、前処理用のパイプラインを定義するdata_processingファイルが作成されます。
kedro pipeline create data_processing
すると、.boston-pls/src/boston_pls/に以下のファイルが作成されます。
.boston-pls/src/boston_pls/
├── pipelines
│ ├── __init__.py
│ ├── data_processing
│ │ ├── README.md
│ │ ├── __init__.py
│ │ ├── __pycache__
│ │ ├── nodes.py
│ │ └── pipeline.py
nodes.pyにデータ前処理用の関数を以下のように定義します。
import pandas as pd
from sklearn.model_selection import train_test_split
def parse_data(raw_df):
data = raw_df.drop(['MedHouseVal'], axis=1)
label = raw_df['MedHouseVal']
return data, label
def split_dataset(data, label):
data_train, data_eval, label_train, label_eval = train_test_split(data, label)
data_fit, data_val, label_fit, label_val = train_test_split(data_train, label_train)
return data_fit, label_fit, data_val, label_val, data_eval, label_eval
続いて、pipeline.pyに以下のスクリプトを書きます。これによりnodes.pyで定義した関数を繋いでパイプラインが構築できる。
from kedro.pipeline import Pipeline, node, pipeline
from .nodes import parse_data, split_dataset
def create_pipeline(**kwargs) -> Pipeline:
return pipeline(
[
node(
func=parse_data,
inputs="boston",
outputs=["data", "label"],
name="parse_data",
),
node(
func=split_dataset,
inputs=["data", "label"],
outputs=["data_fit", "label_fit","data_val", "label_val", "data_eval", "label_eval"],
name="split_dataset",
),
]
)
続いて、以下のコマンドを実行し、モデル学習用のパイプラインを定義するdata_scienceファイルが作成されます。
kedro pipeline create data_science
すると、.boston-pls/src/boston_pls/に以下のファイルが作成されます。
.boston-pls/src/boston_pls/
├── pipelines
│ ├── __init__.py
│ ├── data_science
│ │ ├── README.md
│ │ ├── __init__.py
│ │ ├── __pycache__
│ │ ├── nodes.py
│ │ └── pipeline.py
nodes.pyに学習用の関数を以下のように定義します。
import mlflow
import pandas as pd
import matplotlib.pyplot as plt
import numpy as np
import os
import shutil
import pickle
from sklearn.cross_decomposition import PLSRegression
from sklearn.metrics import r2_score
import seaborn as sns
def data_prepare(data_fit,label_fit, data_val, label_val, data_eval, label_eval):
data_fit_scaled = (data_fit - data_fit.min()) / (data_fit.max() - data_fit.min())
data_val_scaled = (data_val - data_fit.min()) / (data_fit.max() - data_fit.min())
data_eval_scaled = (data_eval - data_fit.min()) / (data_fit.max() - data_fit.min())
label_fit_scaled = (label_fit - label_fit.mean()) / label_fit.std()
label_val_scaled = (label_val - label_fit.mean()) / label_fit.std()
label_eval_scaled = (label_eval - label_fit.mean()) / label_fit.std()
return data_fit_scaled, data_val_scaled, data_eval_scaled, label_fit_scaled, label_val_scaled, label_eval_scaled
def train_model(data_fit_scaled, label_fit, label_fit_scaled, data_val_scaled, label_val, label_val_scaled):
experiment_id = mlflow.create_experiment('boston-pls')
max_pls_component_number = data_fit_scaled.shape[1]
pls_components = np.arange(1, min(np.linalg.matrix_rank(data_fit_scaled) + 1, max_pls_component_number + 1), 1)
with mlflow.start_run(experiment_id=experiment_id):
r2_train_all = list()
r2_val_all = list()
for pls_component in pls_components:
with mlflow.start_run(nested=True, experiment_id=experiment_id):
pls_model = PLSRegression(n_components=pls_component)
pls_model.fit(data_fit_scaled, label_fit_scaled)
train_pred_scaled = pls_model.predict(data_fit_scaled)
val_pred_scaled = pls_model.predict(data_val_scaled)
train_pred = train_pred_scaled * label_fit.std(ddof=1) + label_fit.mean()
val_pred = val_pred_scaled * label_fit.std(ddof=1) + label_fit.mean()
r2_train = r2_score(label_fit, train_pred)
r2_val = r2_score(label_val, val_pred)
r2_train_all.append(r2_train)
r2_val_all.append(r2_val)
""" MLFlow管理"""
# ハイパーパラメータ, 評価指標, 学習済みモデルをMLflowへ保存
mlflow.log_param("n_components", pls_component)
mlflow.log_metric("R2_train", r2_train)
mlflow.log_metric("R2_val", r2_val)
mlflow.sklearn.log_model(pls_model, "model")
optimal_pls_component_number = np.where(r2_val_all == np.max(r2_val_all))
optimal_pls_component_number = optimal_pls_component_number[0][0] + 1
best_model = PLSRegression(n_components=optimal_pls_component_number)
best_model.fit(data_fit_scaled, label_fit_scaled)
return experiment_id, r2_train_all, r2_val_all, best_model
def dump_study(experiment_id, r2_train_all, r2_val_all, best_model, data_fit, label_fit, data_val, label_val):
sns.set()
max_pls_component_number = data_fit.shape[1]
pls_components = np.arange(1, min(np.linalg.matrix_rank(data_fit) + 1, max_pls_component_number + 1), 1)
with mlflow.start_run(experiment_id = experiment_id):
os.makedirs("results", exist_ok=True)
plt.figure(figsize=(12,6))
plt.title('Boston-PLS')
plt.plot(pls_components, r2_train_all, label='train')
plt.plot(pls_components, r2_val_all,label='val')
plt.xlabel('N_components')
plt.ylabel('R2')
plt.legend()
plt.savefig(os.path.join("results", "r2_score.png"))
plt.show()
data_fit.to_csv(os.path.join("results", "train_feature.csv"))
label_fit.to_csv(os.path.join("results", "train_label.csv"))
data_val.to_csv(os.path.join("results", "val_feature.csv"))
label_val.to_csv(os.path.join("results", "val_label.csv"))
#pls-vip法による変数重要度の可視化
plt.figure(figsize=(12,6))
sel_var , vips = _pls_vip(best_model.x_weights_, best_model.y_loadings_.T, best_model.x_scores_ )
vips_df = pd.DataFrame(vips, index=data_fit.columns, columns=['vips']).sort_values('vips', ascending=False)
plt.barh(vips_df.index, vips_df['vips'])
plt.savefig(os.path.join("results", "vips.png"))
with open(os.path.join("results", "model.picke"), "wb") as f:
pickle.dump(best_model, f, protocol=2)
mlflow.log_artifacts("results", artifact_path="results")
shutil.rmtree("results")
def eval_model(experiment_id, best_model,label_fit, data_eval_scaled, label_eval):
with mlflow.start_run(experiment_id=experiment_id):
eval_pred_scaled = best_model.predict(data_eval_scaled)
eval_pred = eval_pred_scaled * label_fit.std(ddof=1) + label_fit.mean()
r2_eval = r2_score(label_eval, eval_pred)
mlflow.log_metric("R2_test_best_model", r2_eval)
#PLS特徴量重要度の可視化
def _pls_vip(W, D, T):
M, R = W.shape
weight = np.zeros([R])
vips = np.zeros([M])
ssy = np.diag(T.T @ T @ D @ D.T)
total_ssy = np.sum(ssy)
for m in range(M):
for r in range(R):
weight[r] = np.array([(W[m, r]/ np.linalg.norm(W[:, r]))**2])
vips[m] = np.sqrt(M*(ssy @ weight) / total_ssy)
sel_var = np.arange(M)
sel_var = sel_var[vips>=1]
return sel_var, vips
回帰モデルとしてPLSを採用し、パイプラインの中にPLSのハイパーパラメータである圧縮次元サイズを変えてモデルを作成し、圧縮次元サイズとモデル予測精度の関係をMLFlowに保存する機能を組み込む。
最後にpipeline.pyに以下のスクリプトを追加してパイプラインを完成させます。
from kedro.pipeline import Pipeline, node, pipeline
from .nodes import *
def create_pipeline(**kwargs) -> Pipeline:
return pipeline(
[
node(
func=data_prepare,
inputs=["data_fit","label_fit", "data_val", "label_val", "data_eval", "label_eval"],
outputs=["data_fit_scaled", "data_val_scaled", "data_eval_scaled", "label_fit_scaled", "label_val_scaled", "label_eval_scaled"],
name="data_prepare",
),
node(
func=train_model,
inputs=["data_fit_scaled", "label_fit", "label_fit_scaled", "data_val_scaled", "label_val", "label_val_scaled"],
outputs=["exp_id", "r2_train", "r2_val", "best_model"],
name="train_model",
),
node(
func=dump_study,
inputs=["exp_id", "r2_train", "r2_val", "best_model", "data_fit", "label_fit", "data_val", "label_val"],
outputs=None,
name="dump_study",
),
node(
func=eval_model,
inputs=["exp_id", "best_model","label_fit", "data_eval_scaled", "label_eval"],
outputs=None,
name="eval_model",
)
]
)
以上で準備は完了です。./boston-pls/src/boston-pls/で以下のコマンドを実行すると設定したパイプラインで学習が行われます。
パイプラインの実行
kedro run
特にエラーがなければ以下のログがコマンドライン上に表示され、完了します。
[02/28/23 23:31:43] INFO Kedro project boston-pls session.py:355
[02/28/23 23:31:45] INFO Loading data from 'boston' (CSVDataSet)... data_catalog.py:343
INFO Running node: parse_data: parse_data([boston]) -> [data,label] node.py:329
INFO Saving data to 'data' (MemoryDataSet)... data_catalog.py:382
INFO Saving data to 'label' (MemoryDataSet)... data_catalog.py:382
INFO Completed 1 out of 6 tasks sequential_runner.py:85
INFO Loading data from 'data' (MemoryDataSet)... data_catalog.py:343
INFO Loading data from 'label' (MemoryDataSet)... data_catalog.py:343
INFO Running node: split_dataset: split_dataset([data,label]) -> node.py:329
[data_fit,label_fit,data_val,label_val,data_eval,label_eval]
INFO Saving data to 'data_fit' (MemoryDataSet)... data_catalog.py:382
INFO Saving data to 'label_fit' (MemoryDataSet)... data_catalog.py:382
INFO Saving data to 'data_val' (MemoryDataSet)... data_catalog.py:382
INFO Saving data to 'label_val' (MemoryDataSet)... data_catalog.py:382
INFO Saving data to 'data_eval' (MemoryDataSet)... data_catalog.py:382
INFO Saving data to 'label_eval' (MemoryDataSet)... data_catalog.py:382
INFO Completed 2 out of 6 tasks sequential_runner.py:85
INFO Loading data from 'data_fit' (MemoryDataSet)... data_catalog.py:343
INFO Loading data from 'label_fit' (MemoryDataSet)... data_catalog.py:343
INFO Loading data from 'data_val' (MemoryDataSet)... data_catalog.py:343
INFO Loading data from 'label_val' (MemoryDataSet)... data_catalog.py:343
INFO Loading data from 'data_eval' (MemoryDataSet)... data_catalog.py:343
INFO Loading data from 'label_eval' (MemoryDataSet)... data_catalog.py:343
INFO Running node: data_prepare: node.py:329
data_prepare([data_fit,label_fit,data_val,label_val,data_eval,label_eval]) ->
[data_fit_scaled,data_val_scaled,data_eval_scaled,label_fit_scaled,label_val_scale
d,label_eval_scaled]
INFO Saving data to 'data_fit_scaled' (MemoryDataSet)... data_catalog.py:382
INFO Saving data to 'data_val_scaled' (MemoryDataSet)... data_catalog.py:382
INFO Saving data to 'data_eval_scaled' (MemoryDataSet)... data_catalog.py:382
INFO Saving data to 'label_fit_scaled' (MemoryDataSet)... data_catalog.py:382
INFO Saving data to 'label_val_scaled' (MemoryDataSet)... data_catalog.py:382
INFO Saving data to 'label_eval_scaled' (MemoryDataSet)... data_catalog.py:382
INFO Completed 3 out of 6 tasks sequential_runner.py:85
INFO Loading data from 'data_fit_scaled' (MemoryDataSet)... data_catalog.py:343
INFO Loading data from 'label_fit' (MemoryDataSet)... data_catalog.py:343
INFO Loading data from 'label_fit_scaled' (MemoryDataSet)... data_catalog.py:343
INFO Loading data from 'data_val_scaled' (MemoryDataSet)... data_catalog.py:343
INFO Loading data from 'label_val' (MemoryDataSet)... data_catalog.py:343
INFO Loading data from 'label_val_scaled' (MemoryDataSet)... data_catalog.py:343
INFO Running node: train_model: node.py:329
train_model([data_fit_scaled,label_fit,label_fit_scaled,data_val_scaled,label_val,
label_val_scaled]) -> [exp_id,r2_train,r2_val,best_model]
[02/28/23 23:31:55] INFO Saving data to 'exp_id' (MemoryDataSet)... data_catalog.py:382
INFO Saving data to 'r2_train' (MemoryDataSet)... data_catalog.py:382
INFO Saving data to 'r2_val' (MemoryDataSet)... data_catalog.py:382
INFO Saving data to 'best_model' (MemoryDataSet)... data_catalog.py:382
INFO Completed 4 out of 6 tasks sequential_runner.py:85
INFO Loading data from 'exp_id' (MemoryDataSet)... data_catalog.py:343
INFO Loading data from 'r2_train' (MemoryDataSet)... data_catalog.py:343
INFO Loading data from 'r2_val' (MemoryDataSet)... data_catalog.py:343
INFO Loading data from 'best_model' (MemoryDataSet)... data_catalog.py:343
INFO Loading data from 'data_fit' (MemoryDataSet)... data_catalog.py:343
INFO Loading data from 'label_fit' (MemoryDataSet)... data_catalog.py:343
INFO Loading data from 'data_val' (MemoryDataSet)... data_catalog.py:343
INFO Loading data from 'label_val' (MemoryDataSet)... data_catalog.py:343
INFO Running node: dump_study: node.py:329
dump_study([exp_id,r2_train,r2_val,best_model,data_fit,label_fit,data_val,label_va
l]) -> None
[02/28/23 23:31:56] INFO Completed 5 out of 6 tasks sequential_runner.py:85
INFO Loading data from 'exp_id' (MemoryDataSet)... data_catalog.py:343
INFO Loading data from 'best_model' (MemoryDataSet)... data_catalog.py:343
INFO Loading data from 'label_fit' (MemoryDataSet)... data_catalog.py:343
INFO Loading data from 'data_eval_scaled' (MemoryDataSet)... data_catalog.py:343
INFO Loading data from 'label_eval' (MemoryDataSet)... data_catalog.py:343
INFO Running node: eval_model: node.py:329
eval_model([exp_id,best_model,label_fit,data_eval_scaled,label_eval]) -> None
INFO Completed 6 out of 6 tasks sequential_runner.py:85
INFO Pipeline execution completed successfully. runner.py:93
INFO Loading data from 'label_eval_scaled' (MemoryDataSet)... data_catalog.py:343
また、./src/bosto-pls/にmlrunsというフォルダが作成されます。ここに.src/boston-pls/pipelines/data_processing/node.py中で定義したmlflow実験管理データ(モデルやr2スコア)が格納されます。
./boston-pls/mlruns/
├── 0
│ └── meta.yaml
└── 532802700296094184
├── 2af6eb9ad10b44b19f6b337f9542c66b
├── 30a58760b0cd4268b9ec2477f4f4cf22
├── 30f65f75498b4bdfb7a8d4f867e3c31b
├── 5c9cb141070e422eb899dfa36e6e0363
├── 6af57d0dfca4419989650f705f5d1f5d
├── 70d0e5a34596418ca1817924b4f49704
├── 748f57d1a2f24b1b9ca96ccbb6db7fde
├── cf9db618de3f471d9c376326bfd1e2c1
├── e98200c1a5df46889e2924628d938c89
├── ea47e333b8e9432ca892d32f6c5fb9e0
├── f67bad881cb841b4bb14dc7a170e6205
├── fc5c7e7f81d9460ea9da594a16ae659d
└── meta.yaml
デフォルトでは、実験IDは0に設定されており、指定しなければ実験IDが0のディレクトリの下にRUNIDが追加され、そのRUNIDに対応するモデルとそのモデルの設定ハイパーパラメータ、予測精度が保存されていく。
今回は、デフォルトの実験IDではなく、新しく実験ID(=bosto-pls)を定義しているため、それに紐づく532802700296094184というディレクトが作成され、その下にそれぞれのモデルのデータが保存されている。
./boston-pls/mlruns/532802700296094184/2af6eb9ad10b44b19f6b337f9542c66b/
├── artifacts
│ └── model
│ ├── MLmodel
│ ├── conda.yaml
│ ├── model.pkl
│ ├── python_env.yaml
│ └── requirements.txt
├── meta.yaml
├── metrics
│ ├── R2_train
│ └── R2_val
├── params
│ └── n_components
└── tags
├── mlflow.log-model.history
├── mlflow.parentRunId
├── mlflow.runName
├── mlflow.source.name
├── mlflow.source.type
└── mlflow.user
モデルはartifacts/modelに保存される。またモデルの精度、ハイパーパラメータはそれぞれmetrics/、params/に格納されていることが確認できる。
実験データを閲覧する
mlflowではこのmlrunsファイル内に保存されている実験結果をブラウザ上で確認できる。
./boston-pls/で以下のコマンドを実行します。
mlflow ui (--port 5000)
ポートを指定する場合は--port ポート番号を追加します。
すると以下のブラウザが立ち上がります。

初期起動時は左上のExperimentsのチェックがDefaultに入っているので、boston-plsに変更します。
plsのハイパーパラメータを変えて実験をおこなった結果がRunName:gregarious-hare-668以下に保存されているので展開し、さらにColumnsを開き、全てにチェックを入れます。

すると、各モデルに対する学習データ、テストデータr2スコアが表示されます。今回の場合、plsの圧縮サイズ(=n_componerts)は5が最もバリデーションデータに対する精度が良いことが確認できます。
さらに、Run Name内のリンクをクリックすると、その試行で設定したハイパーパラメータ(Paramtersタブ)、予測精度(Metricsタブ)、学習済みモデル等(Artifactsタブ)が格納されたページに遷移します。
Artifactsタブを展開すると、以下のように格納されている学習済モデルファイルを呼び出すpythonスクリプトが表示されます。
このスクリプトを実行することで、学習済みモデルを使用して回帰をすることができます。

6. 最後に
今回、kedro + MLFlowを使って機械学習モデル実験管理パイプラインを作ってみました。
MLOpsパイプライン(Kedro + MLFlow)作成の参考になれば幸いです。


